这取决于经验实现和并行化
如果您在Numpy中使用Intel SVML,请在其他软件包(如Numba,Numexpr或Cython)中也使用它。 Numba性能提示
如果Numpy命令已经并行化,请尝试在Numba或Cython中并行化它。
代码
import os
os.environ["MKL_NUM_THREADS"] = "1"
import numpy as np
from llvmlite import binding
binding.set_option('SVML', '-vector-library=SVML')
import numba as nb
def py_expsum(x):
return np.sum( np.exp(x) )
@nb.njit(parallel=False,fastmath=True)
def nb_expsum(x):
val = nb.float32(0.)
for ix in nb.prange(x.shape[0]):
for iy in range(x.shape[1]):
val += np.exp(x[ix,iy])
return val
N,M=2000, 1000
a=np.random.rand(N*M).reshape((N,M))
基准测试
#float64
%timeit py_expsum(a) #os.environ["MKL_NUM_THREADS"] = "1"
#7.44 ms ± 86.7 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
%timeit py_expsum(a) #os.environ["MKL_NUM_THREADS"] = "6"
#4.83 ms ± 139 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
%timeit nb_expsum(a) #parallel=false
#2.49 ms ± 25.1 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
%timeit nb_expsum(a) ##parallel=true
#568 µs ± 45.2 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
#float32
%timeit py_expsum(a) #os.environ["MKL_NUM_THREADS"] = "1"
#3.44 ms ± 66.7 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
%timeit py_expsum(a) #os.environ["MKL_NUM_THREADS"] = "6"
#2.59 ms ± 35.7 µs per loop (mean ± std. dev. of 7 runs, 100 loops each)
%timeit nb_expsum(a) #parallel=false
#1 ms ± 12.6 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
%timeit nb_expsum(a) #parallel=true
#252 µs ± 19.5 µs per loop (mean ± std. dev. of 7 runs, 1000 loops each)
使用SVML进行性能绘图
import numpy as np
from llvmlite import binding
binding.set_option('SVML', '-vector-library=SVML')
import numba as nb
def py_expsum(x):
return np.sum(np.exp(x))
@nb.jit( nopython=True,parallel=False,fastmath=False)
def nb_expsum_single_thread(x):
nx, ny = x.shape
val = 0.0
for ix in range(nx):
for iy in range(ny):
val += np.exp( x[ix, iy] )
return val
@nb.jit( nopython=True,parallel=False,fastmath=True)
def nb_expsum_single_thread_vec(x):
nx, ny = x.shape
val = 0.0
for ix in range(nx):
for iy in range(ny):
val += np.exp( x[ix, iy] )
return val
@nb.jit(nopython=True,parallel=True,fastmath=False)
def nb_expsum_parallel(x):
nx, ny = x.shape
val = 0.0
for ix in range(nx):
for iy in nb.prange(ny):
val += np.exp( x[ix, iy] )
return val
@nb.jit(nopython=True,parallel=True,fastmath=True)
def nb_expsum_parallel_vec(x):
nx, ny = x.shape
val = 0.0
for ix in range(nx):
for iy in nb.prange(ny):
val += np.exp( x[ix, iy] )
return val
import perfplot
factor = 1.0
perfplot.show(
setup=lambda n: factor*np.random.rand(1,n),
n_range=[2**k for k in range(0,27)],
kernels=[
py_expsum,
nb_expsum_single_thread,
nb_expsum_single_thread_vec,
nb_expsum_parallel,
nb_expsum_parallel_vec,
cy_expsum
],
logx=True,
logy=True,
xlabel='len(x)'
)
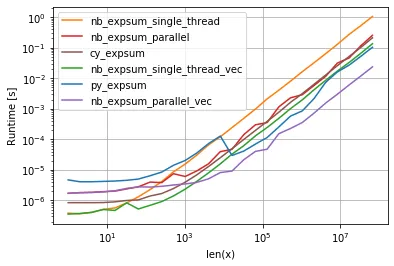
检查是否使用了SVML
可以用来检查一切是否正常运行。
def check_SVML(func):
if 'intel_svmlcc' in func.inspect_llvm(func.signatures[0]):
print("found")
else:
print("not found")
check_SVML(nb_expsum_parallel_vec)
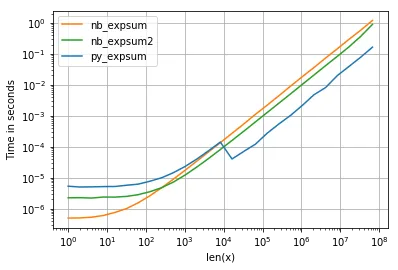
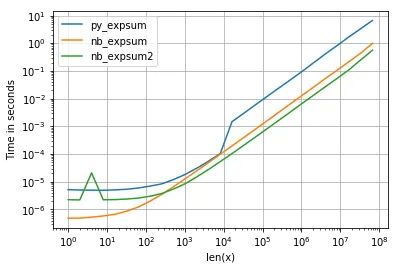
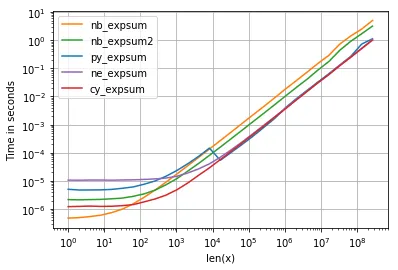

math.exp而不是np.exp。 - Divakarne.evaluate('sum(exp(x))')。 - Brenlla