Numpy提供了
正如这个SO-post所指出的那样,
然而,
考虑以下变体:
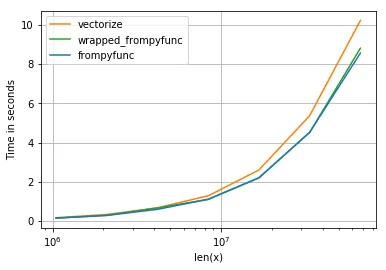
"wrapped_frompyfunc只是将frompyfunc的结果转换为正确的类型 - 我们可以看到,这个操作的成本几乎可以忽略不计。它产生了以下时间(蓝线是frompyfunc):" 我会预期`vectorize`有更多的开销——但这只有在小规模时才能看到。另一方面,将`np.object`转换为`np.float64`也是在`wrapped_frompyfunc`中完成的——但仍然比较快。
这种性能差异如何解释?
使用perfplot包生成时间比较的代码(给定上述函数):
注意:对于更小的尺寸,
vectorize和frompyfunc,它们具有类似的功能。正如这个SO-post所指出的那样,
vectorize封装了frompyfunc并正确处理返回数组的类型,而frompyfunc返回一个np.object类型的数组。然而,
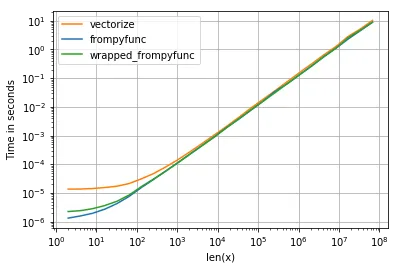
frompyfunc在所有大小的情况下始终比vectorize快10-20%,这也不能用不同的返回类型来解释。考虑以下变体:
import numpy as np
def do_double(x):
return 2.0*x
vectorize = np.vectorize(do_double)
frompyfunc = np.frompyfunc(do_double, 1, 1)
def wrapped_frompyfunc(arr):
return frompyfunc(arr).astype(np.float64)
"wrapped_frompyfunc只是将frompyfunc的结果转换为正确的类型 - 我们可以看到,这个操作的成本几乎可以忽略不计。它产生了以下时间(蓝线是frompyfunc):" 我会预期`vectorize`有更多的开销——但这只有在小规模时才能看到。另一方面,将`np.object`转换为`np.float64`也是在`wrapped_frompyfunc`中完成的——但仍然比较快。
这种性能差异如何解释?
使用perfplot包生成时间比较的代码(给定上述函数):
import numpy as np
import perfplot
perfplot.show(
setup=lambda n: np.linspace(0, 1, n),
n_range=[2**k for k in range(20,27)],
kernels=[
frompyfunc,
vectorize,
wrapped_frompyfunc,
],
labels=["frompyfunc", "vectorize", "wrapped_frompyfunc"],
logx=True,
logy=False,
xlabel='len(x)',
equality_check = None,
)
注意:对于更小的尺寸,
vectorize 的开销要高得多,但这是可以预料的(毕竟它包装了 frompyfunc):
注意事项
vectorize函数主要是为了方便而提供的,而不是为了性能。实现本质上是一个for循环。 如果未指定otypes,则将使用带有第一个参数的函数调用来确定输出的数量。如果cache为True,则会缓存此调用的结果,以防止多次调用该函数。然而,为了实现缓存,必须包装原始函数,这将减慢后续调用的速度,因此只有在您的函数很耗时时才这样做。 - Giacomo Alzettavectorize包装了frompyfunc和 …*”,我觉得很明显vectorize更慢。vectorize(1)调用frompyfunc并且另外(2)做一些其他的事情。为什么你期望做(1)+(2)比只做(2)更快呢? - Socowiwrapped_frompyfunc进行 (2) 操作,而且它比vectorize要快得多。 - eadvectorize所做的相提并论。它也无法与vectorize所带来的额外开销相比。当然,这个例子比vectorize更快。 - user3483203vectorize代码。仅凭推理和时间不足够。 - hpaulj