我刚遇到了相同的问题。
简短回答
使用 InterpolatedUnivariateSpline 代替:
f = InterpolatedUnivariateSpline(row1, row2)
return f(interp)
长回答
UnivariateSpline是一种“对给定数据点进行一维平滑样条拟合”的方法,而InterpolatedUnivariateSpline是一种“给定数据点的一维插值样条”。前者平滑数据,而后者则是一种更传统的插值方法,并且产生了与interp1d期望的结果相同。下图说明了它们之间的区别。
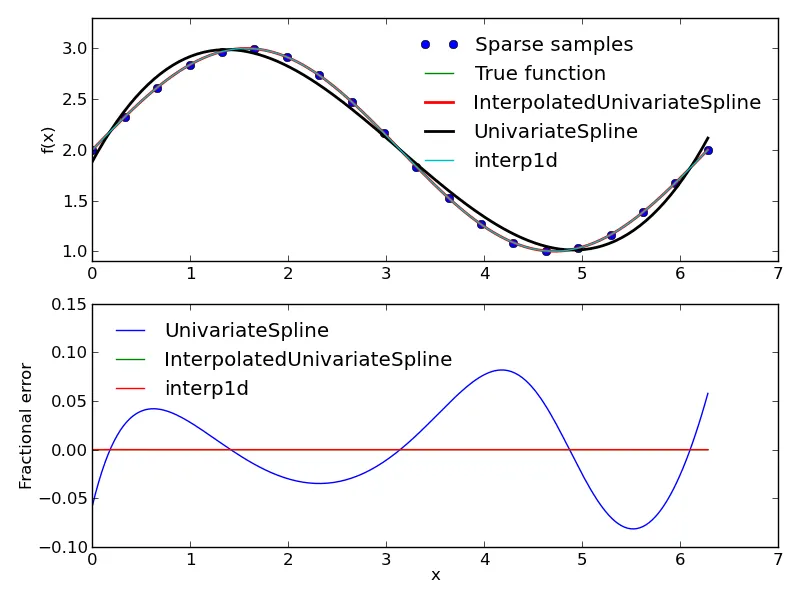
以下是复制该图像所需的代码。
import scipy.interpolate as ip
sparse = linspace(0, 2 * pi, num = 20)
dense = linspace(0, 2 * pi, num = 200)
f = lambda x: sin(x) + 2
fsparse = f(sparse)
fdense = f(dense)
ax = subplot(2, 1, 1)
plot(sparse, fsparse, label = 'Sparse samples', linestyle = 'None', marker = 'o')
plot(dense, fdense, label = 'True function')
interpolate = ip.InterpolatedUnivariateSpline(sparse, fsparse)
plot(dense, interpolate(dense), label = 'InterpolatedUnivariateSpline', linewidth = 2)
smoothing = ip.UnivariateSpline(sparse, fsparse)
plot(dense, smoothing(dense), label = 'UnivariateSpline', color = 'k', linewidth = 2)
ip1d = ip.interp1d(sparse, fsparse, kind = 'cubic')
plot(dense, ip1d(dense), label = 'interp1d')
ylim(.9, 3.3)
legend(loc = 'upper right', frameon = False)
ylabel('f(x)')
subplot(2, 1, 2, sharex = ax)
plot(dense, smoothing(dense) / fdense - 1, label = 'UnivariateSpline')
plot(dense, interpolate(dense) / fdense - 1, label = 'InterpolatedUnivariateSpline')
plot(dense, ip1d(dense) / fdense - 1, label = 'interp1d')
ylabel('Fractional error')
xlabel('x')
ylim(-.1,.15)
legend(loc = 'upper left', frameon = False)
tight_layout()