在跟踪线性函数的案例后,我认为可以采用类似的方法。不过,解决拉格朗日问题似乎非常繁琐,当然也是可能的。我想出了一种似乎合理且应该能够得到非常相似结果的不同测量方法。我重新调整误差椭圆,使其成为一个切点。我以这个接触点(X_k,Y_k)到距离作为卡方的度量,它是从(x_k-X_k/sx_k)**2+(y_k-Y_k/sy_k)**2计算出来的。因为在纯y误差的情况下,这是标准的最小二乘拟合。对于纯x误差,它只是反过来。对于等于的x,y误差,它将给出垂直规则,即最短距离。使用相应的卡方函数scipy.optimize.leastsq已经提供了从Hesse近似的协方差矩阵。但需要注意的是,参数之间存在强相关性。
我的过程如下:
import matplotlib
matplotlib.use('Qt5Agg')
import matplotlib.pyplot as plt
import numpy as np
import myModules.multipoleMoments as mm
from random import random
from scipy.optimize import minimize,leastsq
def boxmuller(x0,sigma):
u1=random()
u2=random()
ll=np.sqrt(-2*np.log(u1))
z0=ll*np.cos(2*np.pi*u2)
z1=ll*np.cos(2*np.pi*u2)
return sigma*z0+x0, sigma*z1+x0
def f(t,c,d,n):
return c+d*np.abs(t)**n
def test_data(c,d,n, xList,const_sx,rel_sx,const_sy,rel_sy):
yList=[f(t,c,d,n) for t in xList]
xErrList=[ boxmuller(x,const_sx+x*rel_sx)[0] for x in xList]
yErrList=[ boxmuller(y,const_sy+y*rel_sy)[0] for y in yList]
return xErrList,yErrList
def elliptic_rescale(x,c,d,n,x0,y0,sa,sb):
y=f(x,c,d,n)
r=np.sqrt((x-x0)**2+(y-y0)**2)
kappa=float(sa)/float(sb)
tau=np.arctan2(y-y0,x-x0)
new_a=r*np.sqrt(np.cos(tau)**2+(kappa*np.sin(tau))**2)
return new_a
def ell_data(a,b,x0=0,y0=0):
tList=np.linspace(0,2*np.pi,150)
k=float(a)/float(b)
rList=[a/np.sqrt((np.cos(t))**2+(k*np.sin(t))**2) for t in tList]
xyList=np.array([[x0+r*np.cos(t),y0+r*np.sin(t)] for t,r in zip(tList,rList)])
return xyList
def residuals(parameters,dataPoint):
c,d,n= parameters
theData=np.array(dataPoint)
best_t_List=[]
for i in range(len(dataPoint)):
x,y,sx,sy=dataPoint[i][0],dataPoint[i][1],dataPoint[i][2],dataPoint[i][3]
ed_fit=minimize(elliptic_rescale,0,args=(c,d,n,x,y,sx,sy) )
best_t=ed_fit['x'][0]
best_t_List+=[best_t]
best_y_List=[f(t,c,d,n) for t in best_t_List]
wighted_dx_List=[(x_b-x_f)/sx for x_b,x_f,sx in zip( best_t_List,theData[:,0], theData[:,2] ) ]
wighted_dy_List=[(x_b-x_f)/sx for x_b,x_f,sx in zip( best_y_List,theData[:,1], theData[:,3] ) ]
return wighted_dx_List+wighted_dy_List
cc,dd,nn=2.177,.735,1.75
ssaa,ssbb=1,3
xx0,yy0=2,3
csx,rsx=.1,.05
csy,rsy=.4,.00
xThData=np.linspace(0,3,15)
yThData=[ f(t, cc,dd,nn) for t in xThData]
xNoiseData,yNoiseData=test_data(cc,dd,nn, xThData, csx,rsx, csy,rsy)
xGuessdError=[csx+rsx*x for x in xNoiseData]
yGuessdError=[csy+rsy*y for y in yNoiseData]
fig1 = plt.figure(1)
ax=fig1.add_subplot(1,1,1)
ax.plot(xThData,yThData)
ax.errorbar(xNoiseData,yNoiseData, xerr=xGuessdError, yerr=yGuessdError, fmt='none',ecolor='r')
for i in range(len(xNoiseData)):
el0=ell_data(xGuessdError[i],yGuessdError[i],x0=xNoiseData[i],y0=yNoiseData[i])
ax.plot(*zip(*el0),linewidth=1, color="#808080",linestyle='-')
ed_fit=minimize(elliptic_rescale,0,args=(cc,dd,nn,xNoiseData[i],yNoiseData[i],xGuessdError[i],yGuessdError[i]) )
best_t=ed_fit['x'][0]
best_a=elliptic_rescale(best_t,cc,dd,nn,xNoiseData[i],yNoiseData[i],xGuessdError[i],yGuessdError[i])
el0=ell_data(best_a,best_a/xGuessdError[i]*yGuessdError[i],x0=xNoiseData[i],y0=yNoiseData[i])
ax.plot(*zip(*el0),linewidth=1, color="#a000a0",linestyle='-')
zipData=zip(xNoiseData,yNoiseData, xGuessdError, yGuessdError)
estimate = [2.0,1,2.5]
bestFitValues, cov,info,mesg, ier = leastsq(residuals, estimate,args=(zipData), full_output=1)
print bestFitValues
s_sq = (np.array(residuals(bestFitValues, zipData))**2).sum()/(len(zipData)-len(estimate))
pcov = cov * s_sq
print pcov
ax.plot(xThData,[f(x,*bestFitValues) for x in xThData])
for i in range(len(xNoiseData)):
ed_fit=minimize(elliptic_rescale,0,args=(bestFitValues[0],bestFitValues[1],bestFitValues[2],xNoiseData[i],yNoiseData[i],xGuessdError[i],yGuessdError[i]) )
best_t=ed_fit['x'][0]
best_a=elliptic_rescale(best_t,bestFitValues[0],bestFitValues[1],bestFitValues[2],xNoiseData[i],yNoiseData[i],xGuessdError[i],yGuessdError[i])
el0=ell_data(best_a,best_a/xGuessdError[i]*yGuessdError[i],x0=xNoiseData[i],y0=yNoiseData[i])
ax.plot(*zip(*el0),linewidth=1, color="#f0a000",linestyle='-')
plt.show()
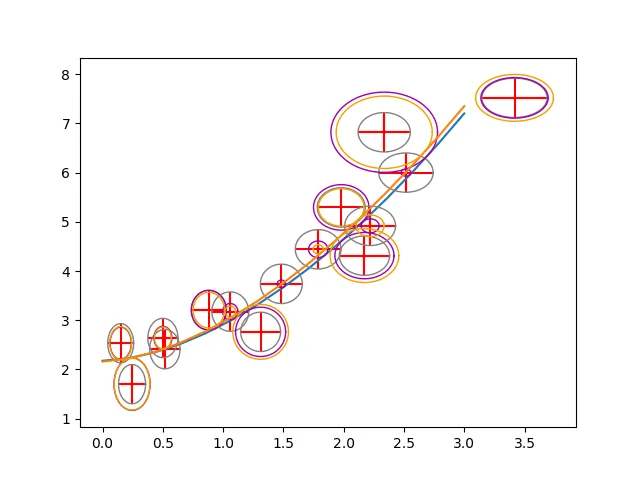 蓝色曲线是原始函数。红色误差条数据点是由此计算出来的。灰色椭圆显示“常数概率密度线”。紫色椭圆以原始图形为切线,橙色椭圆以拟合为切线。
蓝色曲线是原始函数。红色误差条数据点是由此计算出来的。灰色椭圆显示“常数概率密度线”。紫色椭圆以原始图形为切线,橙色椭圆以拟合为切线。
这里的最佳拟合值是(不是您的数据!):
[ 2.16146783 0.80204967 1.69951865]
协方差矩阵的形式为:
[[ 0.0644794 -0.05418743 0.05454876]
[-0.05418743 0.07228771 -0.08172885]
[ 0.05454876 -0.08172885 0.10173394]]
编辑
关于“椭圆距离”,我认为这正是所链接论文中的拉格朗日方法所做的。只是对于直线,你可以写出一个精确解,而在这种情况下你不能。
更新
我遇到了一些与OP数据有关的问题。不过通过重新缩放,它可以工作。由于斜率和指数高度相关,因此我首先必须弄清楚协方差矩阵如何针对重新缩放的数据发生变化。详细信息可以在J. Phys. Chem. 105 (2001) 3917中找到。
使用上面的函数定义,数据处理将如下所示:
cc,dd,nn=.2,1,7.5
csx,rsx=2.0,.0
csy,rsy=0,.5
yNoiseData=np.array([1.71,1.76, 1.81, 1.86, 1.93, 2.01, 2.09, 2.20, 2.32, 2.47, 2.65, 2.87, 3.16, 3.53, 4.02, 4.69, 5.64, 7.07, 9.35,13.34,21.43])
xNoiseData=np.array([0.0,5.0,10.0,15.0,20.0,25.0,30.0,35.0,40.0,45.0,50.0,55.0,60.0,65.0,70.0,75.0,80.0,85.0,90.0,95.0,100.0])
xGuessdError=[csx+rsx*x for x in xNoiseData]
yGuessdError=[csy+rsy*y for y in yNoiseData]
fig1 = plt.figure(1)
ax=fig1.add_subplot(1,2,2)
bx=fig1.add_subplot(1,2,1)
ax.errorbar(xNoiseData,yNoiseData, xerr=xGuessdError, yerr=yGuessdError, fmt='none',ecolor='r')
print "\n++++++++++++++++++++++++ scaled ++++++++++++++++++++++++\n"
alpha=.05
beta=.01
xNoiseDataS = [ beta*x for x in xNoiseData ]
yNoiseDataS = [ alpha*x for x in yNoiseData ]
xGuessdErrorS = [ beta*x for x in xGuessdError ]
yGuessdErrorS = [ alpha*x for x in yGuessdError ]
xtmp=np.linspace(0,1.1,25)
bx.errorbar(xNoiseDataS,yNoiseDataS, xerr=xGuessdErrorS, yerr=yGuessdErrorS, fmt='none',ecolor='r')
zipData=zip(xNoiseDataS,yNoiseDataS, xGuessdErrorS, yGuessdErrorS)
estimate = [.1,1,7.5]
bestFitValues, cov,info,mesg, ier = leastsq(residuals, estimate,args=(zipData), full_output=1)
print bestFitValues
plt.plot(xtmp,[ f(x,*bestFitValues)for x in xtmp])
s_sq = (np.array(residuals(bestFitValues, zipData))**2).sum()/(len(zipData)-len(estimate))
pcov = cov * s_sq
print pcov
print "\n++++++++++++++++++++++++ scaled back ++++++++++++++++++++++++\n"
realBestFitValues= [bestFitValues[0]/alpha, bestFitValues[1]/alpha*(beta)**bestFitValues[2],bestFitValues[2] ]
print realBestFitValues
uMX = np.array( [[1/alpha,0,0],[0,beta**bestFitValues[2]/alpha,bestFitValues[1]/alpha*beta**bestFitValues[2]*np.log(beta)],[0,0,1]] )
uMX_T = uMX.transpose()
realCov = np.dot(uMX, np.dot(pcov,uMX_T))
print realCov
for i,para in enumerate(["b","a","n"]):
print para+" = "+"{:.2e}".format(realBestFitValues[i])+" +/- "+"{:.2e}".format(np.sqrt(realCov[i,i]))
ax.plot(xNoiseData,[f(x,*realBestFitValues) for x in xNoiseData])
plt.show()
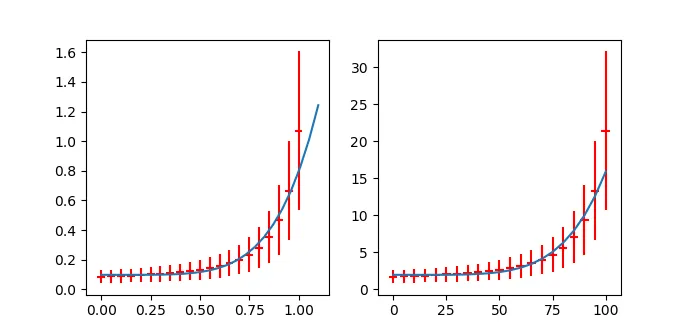
因此,数据已经被合理拟合。然而,我认为还有一个线性项。
输出结果如下:
++++++++++++++++++++++++ scaled ++++++++++++++++++++++++
[ 0.09788886 0.69614911 5.2221032 ]
[[ 1.25914194e-05 2.86541696e-05 6.03957467e-04]
[ 2.86541696e-05 3.88675025e-03 2.00199108e-02]
[ 6.03957467e-04 2.00199108e-02 1.75756532e-01]]
++++++++++++++++++++++++ scaled back ++++++++++++++++++++++++
[1.9577772055183396, 5.0064036934715239e-10, 5.2221031993990517]
[[ 5.03656777e-03 -2.74367539e-11 1.20791493e-02]
[ -2.74367539e-11 8.69854174e-19 -3.90815222e-10]
[ 1.20791493e-02 -3.90815222e-10 1.75756532e-01]]
b = 1.96e+00 +/- 7.10e-02
a = 5.01e-10 +/- 9.33e-10
n = 5.22e+00 +/- 4.19e-01
可以看到协方差矩阵中斜率和指数之间有很强的相关性。同时请注意,斜率的误差非常大。
顺便说一句
使用模型b+a*x**n + e*x,我得到了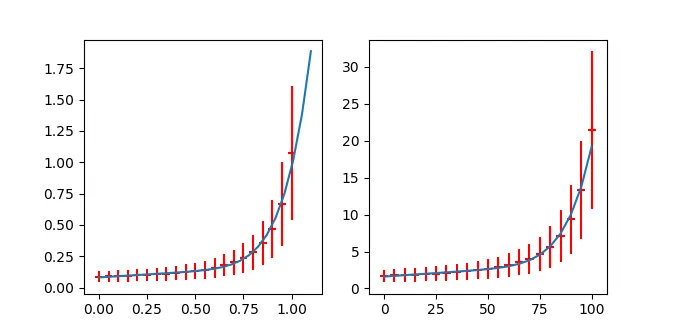 。
。
++++++++++++++++++++++++ scaled ++++++++++++++++++++++++
[ 0.08050174 0.78438855 8.11845402 0.09581568]
[[ 5.96210962e-06 3.07651631e-08 -3.57876577e-04 -1.75228231e-05]
[ 3.07651631e-08 1.39368435e-03 9.85025139e-03 1.83780053e-05]
[ -3.57876577e-04 9.85025139e-03 1.85226736e-01 2.26973118e-03]
[ -1.75228231e-05 1.83780053e-05 2.26973118e-03 7.92853339e-05]]
++++++++++++++++++++++++ scaled back ++++++++++++++++++++++++
[1.6100348667765145, 9.0918698097511416e-16, 8.1184540175879985, 0.019163135651422442]
[[ 2.38484385e-03 2.99690170e-17 -7.15753154e-03 -7.00912926e-05]
[ 2.99690170e-17 3.15340690e-30 -7.64119623e-16 -1.89639468e-18]
[ -7.15753154e-03 -7.64119623e-16 1.85226736e-01 4.53946235e-04]
[ -7.00912926e-05 -1.89639468e-18 4.53946235e-04 3.17141336e-06]]
b = 1.61e+00 +/- 4.88e-02
a = 9.09e-16 +/- 1.78e-15
n = 8.12e+00 +/- 4.30e-01
e = 1.92e-02 +/- 1.78e-03
当然,如果你添加参数,适合度会变得更好,但我认为这在这里看起来还算合理(甚至可能是
b + a*x**n+e*x**m,但这太过深入了)。




scipy的optimize.curve_fit方法没有实现接受unumpy数组。对于这种拟合,您最好使用scikit-learn并使用指数点积(用于实际回归)和白噪声(用于不确定性)内核进行高斯过程回归。我不是专家-尝试一下,如果有问题,请使用scikit-learn标记提问。 - Daniel Funcertainties.wrap与optimize.curve_fit一起工作,但这似乎需要奇迹。不过,为了测试这个问题,改变我在工作中的本地环境是我不愿意经历的痛苦。 - Daniel Fx或仅在y中添加附加误差? 如果是,它在哪里设置? - mikuszefskix= a*y**n+b而不是y= a*x**n+b。在后者中,您没有一个适当的截距。它看起来确实是(x-x0)**(1/n)类型的。顺便问一下,n应该是整数还是反过来看是整数根? - mikuszefski