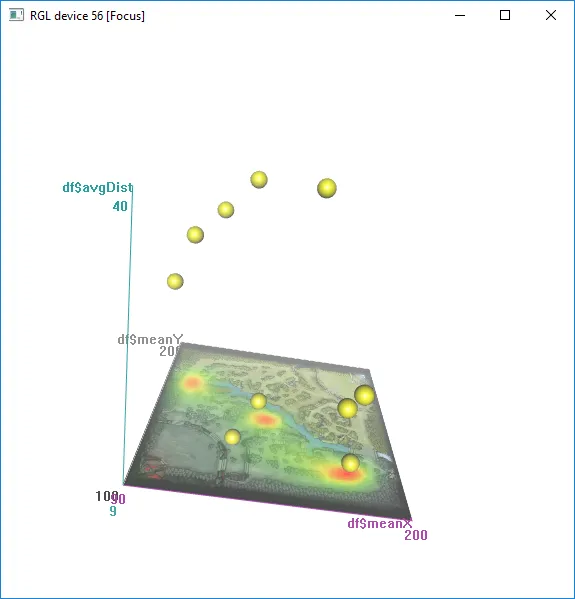
我正在处理三维坐标数据,并在散点图上进行绘制,共有约3万个数据点,此处提供前10个数据以便您复现。
library(rgl)
library(plot3D)
library(car)
df <- data.frame(meanX = c(147.34694,
173.89244,
135.73004,
121.93766,
109.72152,
92.53709,
165.46588,
169.77744,
127.01796,
99.34347),
meanY = c(140.40816,
110.99128,
134.56023,
164.18703,
166.04051,
155.97329,
105.29377,
104.42683,
130.17066,
155.99696),
avgDist = c(40.788118,
12.957329,
14.24348,
39.10424,
34.694258,
25.532335,
21.491695,
23.528944,
9.309201,
31.916879))
我一直在使用scatter3d函数来绘制这个图。
scatter3d(x = df$meanX, y = df$meanY, z = df$avgDist, surface = FALSE)
现在我的“问题”是,我想要一个带有外部图像文件的2D表面覆盖在z = 0处,如果我能够将散点图数据(用于等高线的meanX和meanY)的热力图/等高线投影到该图像上,那就太好了。这是我想要的z = 0的图片:http://i.imgur.com/m6j4q3M.png。这张图片是使用ggplot制作的。
map.colors <- colorRampPalette(c("green","yellow","red"))
densityPlot <- ggplot(direData, aes(x = meanX, y = ,meanY)) +
stat_density2d(geom="tile", aes(fill=..density.., alpha=sqrt(sqrt(..density..))), contour=FALSE, n=100) +
scale_alpha(range = c(0, 1.0)) + scale_fill_gradientn(colours = map.colors(5)) +
xlim(70,185) + ylim(70,185)
minimap <- readPNG('~/yasp/minimap.png')
densityPlot + annotation_raster(minimap, ymin = 70 ,ymax=185 ,xmin = 70,xmax = 185) +
stat_density2d(geom="tile", aes(fill=..density.., alpha=10*sqrt(..density..)), contour=FALSE, n=100)
有没有办法做到这一点?我已经通过谷歌搜索了很多解决方案,但没有找到真正的方法。我不介意首先在ggplot2中创建带有热图的图像,保存它,然后将其用作表面贴合的输入,但如果可以在一个绘图调用中完成所有操作,那当然是非常酷的。