更精确地说,如果将集群解释的方差百分比根据集群数绘制成图形,则前几个集群会添加大量信息(解释大量方差),但在某个点上,边际收益将下降,在图形中形成一个角度。
假设我有以下数据点及其关联的质心,有什么好的方法来计算此度量?
points = numpy.array([[ 0, 0],
[ 0, 1],
[ 0, -1],
[ 1, 0],
[-1, 0],
[ 9, 9],
[ 9, 10],
[ 9, 8],
[10, 9],
[10, 8]])
kmeans(pp,2)
(array([[9, 8],
[0, 0]]), 0.9414213562373096)
我想计算仅给定点和质心的 0.94.. 测量值。不确定是否可以使用Scipy内置方法中的任何方法,还是必须编写自己的方法。对于大量数据,如何高效地进行计算?
简而言之,我的问题(都相关)如下:
- 在给定距离矩阵和将点分配到哪个簇的映射的情况下,如何计算可用于绘制肘部图的度量标准?
- 如果使用不同的距离函数,例如余弦相似性,方法会如何改变?
编辑2:畸变程度(Distortion)
from scipy.spatial.distance import cdist
D = cdist(points, centroids, 'euclidean')
sum(numpy.min(D, axis=1))
第一组点的输出是准确的。然而,当我尝试使用另一组时:
>>> pp = numpy.array([[1,2], [2,1], [2,2], [1,3], [6,7], [6,5], [7,8], [8,8]])
>>> kmeans(pp, 2)
(array([[6, 7],
[1, 2]]), 1.1330618877807475)
>>> centroids = numpy.array([[6,7], [1,2]])
>>> D = cdist(points, centroids, 'euclidean')
>>> sum(numpy.min(D, axis=1))
9.0644951022459797
我猜测最后一个值不匹配的原因是kmeans似乎将该值除以数据集中的总点数。
编辑1:百分比方差
到目前为止,我的代码(应添加到Denis的K-means实现中):
centres, xtoc, dist = kmeanssample( points, 2, nsample=2,
delta=kmdelta, maxiter=kmiter, metric=metric, verbose=0 )
print "Unique clusters: ", set(xtoc)
print ""
cluster_vars = []
for cluster in set(xtoc):
print "Cluster: ", cluster
truthcondition = ([x == cluster for x in xtoc])
distances_inside_cluster = (truthcondition * dist)
indices = [i for i,x in enumerate(truthcondition) if x == True]
final_distances = [distances_inside_cluster[k] for k in indices]
print final_distances
print np.array(final_distances).var()
cluster_vars.append(np.array(final_distances).var())
print ""
print "Sum of variances: ", sum(cluster_vars)
print "Total Variance: ", points.var()
print "Percent: ", (100 * sum(cluster_vars) / points.var())
以下是 k=2 时的输出结果:
Unique clusters: set([0, 1])
Cluster: 0
[1.0, 2.0, 0.0, 1.4142135623730951, 1.0]
0.427451660041
Cluster: 1
[0.0, 1.0, 1.0, 1.0, 1.0]
0.16
Sum of variances: 0.587451660041
Total Variance: 21.1475
Percent: 2.77787757437
在我的真实数据集上(我觉得不对!):
Sum of variances: 0.0188124746402
Total Variance: 0.00313754329764
Percent: 599.592510943
Unique clusters: set([0, 1, 2, 3])
Sum of variances: 0.0255808508714
Total Variance: 0.00313754329764
Percent: 815.314672809
Unique clusters: set([0, 1, 2, 3, 4])
Sum of variances: 0.0588210052519
Total Variance: 0.00313754329764
Percent: 1874.74720416
Unique clusters: set([0, 1, 2, 3, 4, 5])
Sum of variances: 0.0672406353655
Total Variance: 0.00313754329764
Percent: 2143.09824556
Unique clusters: set([0, 1, 2, 3, 4, 5, 6])
Sum of variances: 0.0646291452839
Total Variance: 0.00313754329764
Percent: 2059.86465055
Unique clusters: set([0, 1, 2, 3, 4, 5, 6, 7])
Sum of variances: 0.0817517362176
Total Variance: 0.00313754329764
Percent: 2605.5970695
Unique clusters: set([0, 1, 2, 3, 4, 5, 6, 7, 8])
Sum of variances: 0.0912820650486
Total Variance: 0.00313754329764
Percent: 2909.34837831
Unique clusters: set([0, 1, 2, 3, 4, 5, 6, 7, 8, 9])
Sum of variances: 0.102119601368
Total Variance: 0.00313754329764
Percent: 3254.76309585
Unique clusters: set([0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10])
Sum of variances: 0.125549475536
Total Variance: 0.00313754329764
Percent: 4001.52168834
Unique clusters: set([0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11])
Sum of variances: 0.138469402779
Total Variance: 0.00313754329764
Percent: 4413.30651542
Unique clusters: set([0, 1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12])
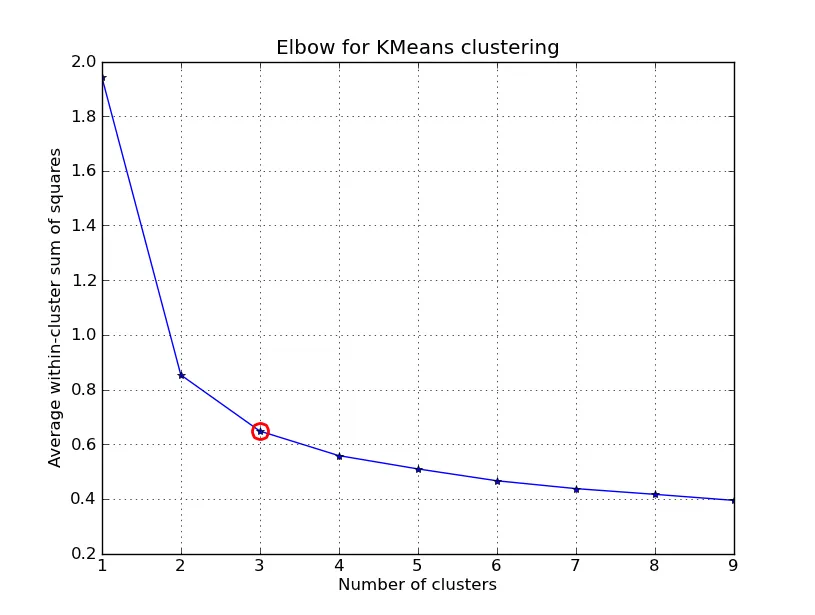
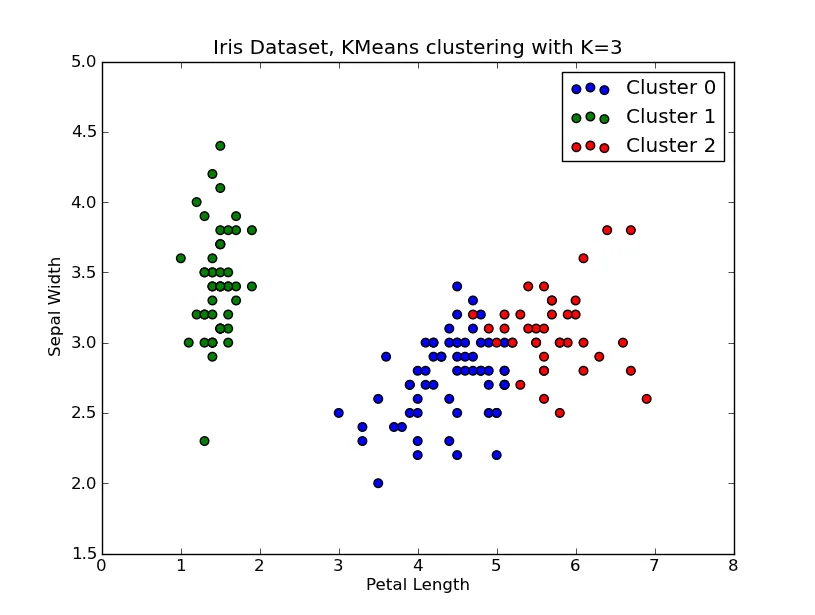
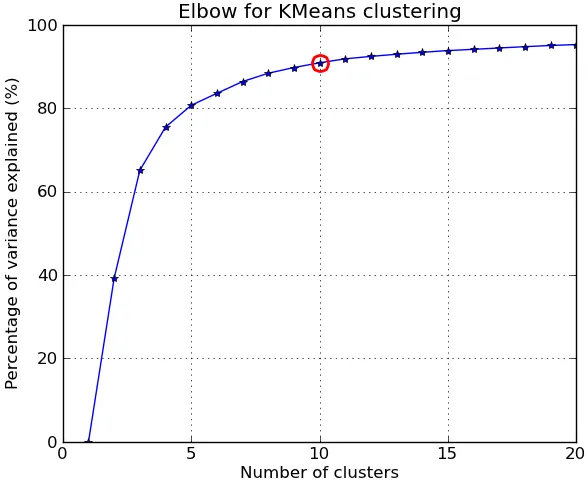

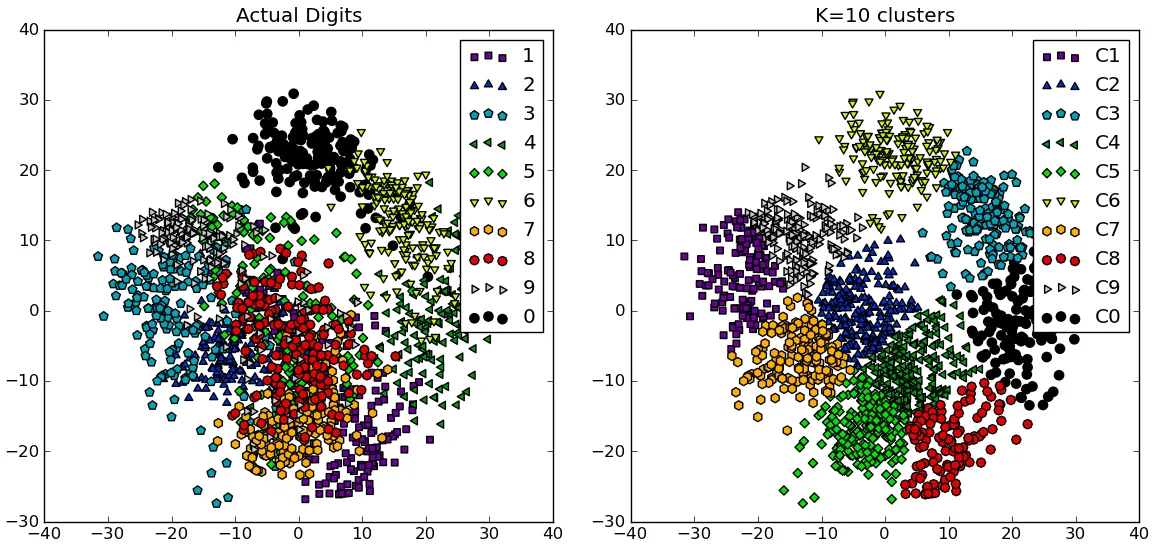
k的价值。在这篇文章中:http://stats.stackexchange.com/questions/9850/how-to-plot-data-output-of-clustering 作者直接使用了畸变值,但我并不真正理解他为什么这样做。您对此有什么想法吗? - Legend