根据文档,参数
我有一些带人工正态分布噪声的数据,这些噪声是变化的:
sigma可用于在拟合中设置数据点的权重。当参数absolute_sigma=True时,它们“描述”1σ误差。我有一些带人工正态分布噪声的数据,这些噪声是变化的:
n = 200
x = np.linspace(1, 20, n)
x0, A, alpha = 12, 3, 3
def f(x, x0, A, alpha):
return A * np.exp(-((x-x0)/alpha)**2)
noise_sigma = x/20
noise = np.random.randn(n) * noise_sigma
yexact = f(x, x0, A, alpha)
y = yexact + noise
如果我想使用curve_fit将带噪声的y拟合到f,那么我应该将sigma设置为什么?文档在这里并不是非常具体,但通常我会使用1/noise_sigma**2作为权重:
p0 = 10, 4, 2
popt, pcov = curve_fit(f, x, y, p0)
popt2, pcov2 = curve_fit(f, x, y, p0, sigma=1/noise_sigma**2, absolute_sigma=True)
然而,它似乎并没有显著提高拟合度。
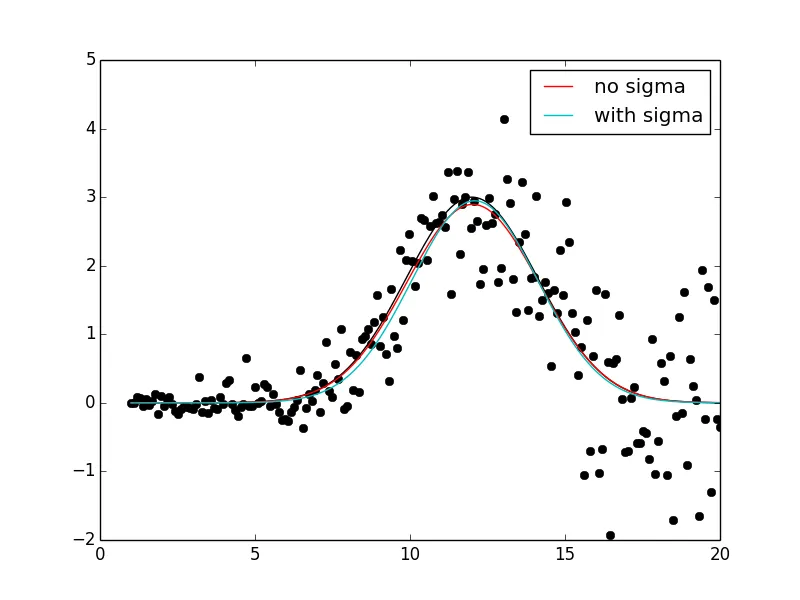
这个选项只是用来通过协方差矩阵更好地解释适应性不确定性的吗?这两者告诉我什么区别?
In [249]: pcov
Out[249]:
array([[ 1.10205238e-02, -3.91494024e-08, 8.81822412e-08],
[ -3.91494024e-08, 1.52660426e-02, -1.05907265e-02],
[ 8.81822412e-08, -1.05907265e-02, 2.20414887e-02]])
In [250]: pcov2
Out[250]:
array([[ 0.26584674, -0.01836064, -0.17867193],
[-0.01836064, 0.27833 , -0.1459469 ],
[-0.17867193, -0.1459469 , 0.38659059]])