我不仅搜索了stackoverflow,还查看了许多其他网站。不幸的是,我还没有找到任何帮助。我将尽可能明确地解释我的问题。由于这是我在stackoverflow上的第一个问题,所以请温柔些,我是R的完全初学者。我的目标是手动向已创建的ggplot2对象添加图例。
这是我正在使用的数据集:
structure(list(Values = 0:5, Count = c(213L, 128L, 37L, 18L,
3L, 1L), rel_freq = c(0.5325, 0.32, 0.0925, 0.045, 0.0075, 0.0025
), pois_distr = c(0.505352031744286, 0.344902761665475, 0.117698067418343,
0.0267763103376731, 0.00456870795136548, 0.000623628635361388
)), class = "data.frame", row.names = c(NA, -6L))
看起来像
Values Count rel_freq pois_distr
1 0 213 0.5325 0.5053520317
2 1 128 0.3200 0.3449027617
3 2 37 0.0925 0.1176980674
4 3 18 0.0450 0.0267763103
5 4 3 0.0075 0.0045687080
6 5 1 0.0025 0.0006236286
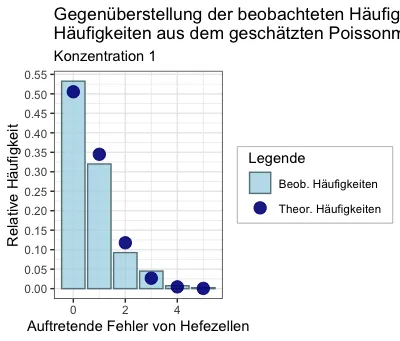
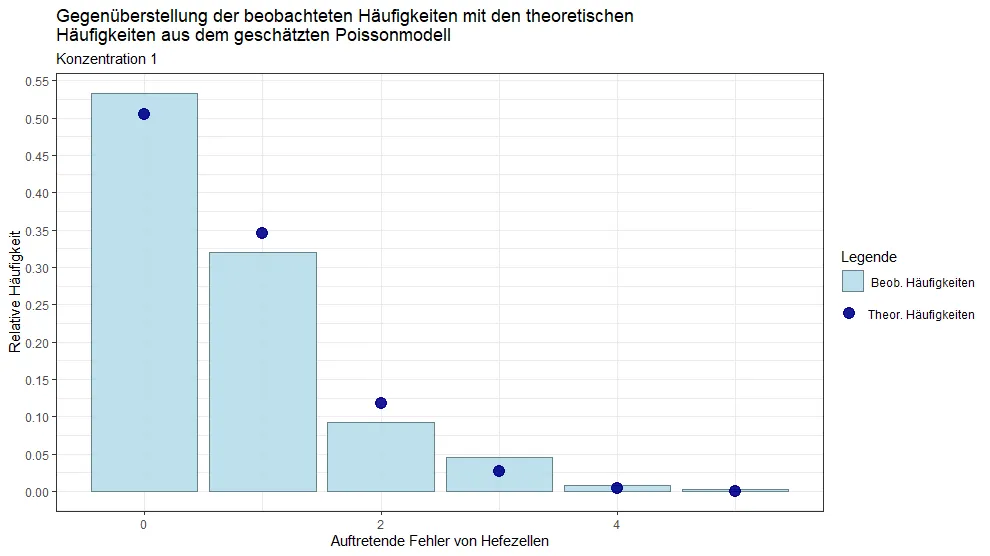
接下来,我已经成功创建了一个ggplot,它还不错,代码如下:
cols <- c('Beob. Häufigkeiten' = 'lightblue', 'Theor. Häufigkeiten' = 'darkblue')
plot_yeast1 <- ggplot(data.frame(data1_plot), aes(x=Values)) +
geom_col(aes(y=rel_freq, fill = 'Beob. Häufigkeiten'), col = 'lightblue4', alpha = 0.8) +
geom_point(aes(y=pois_distr, colour = 'Theor. Häufigkeiten'), alpha = 0.9, size = 4) +
scale_fill_manual(name = 'Legende', values = cols) +
scale_colour_manual(name ='Legende', values = cols) +
scale_y_continuous(breaks = seq(0, 0.6, 0.05)) +
labs(title = 'Gegenüberstellung der beobachteten Häufigkeiten mit den theoretischen \nHäufigkeiten aus dem geschätzten Poissonmodell', x = 'Auftretende Fehler von Hefezellen', y = 'Relative Häufigkeit', subtitle = 'Konzentration 1') +
theme_bw()
plot_yeast1
输出结果为:
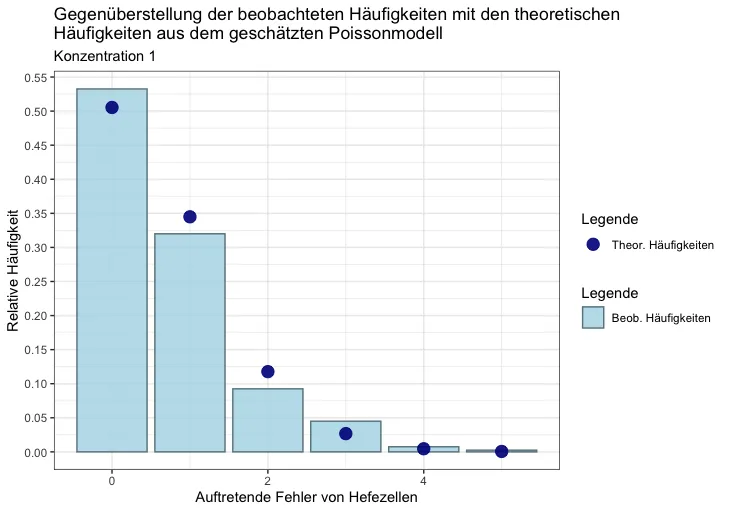
我的目标是将右侧手动创建的两个图例合并成一个。我已经尝试跳过第二个图例的标题,结果如下:
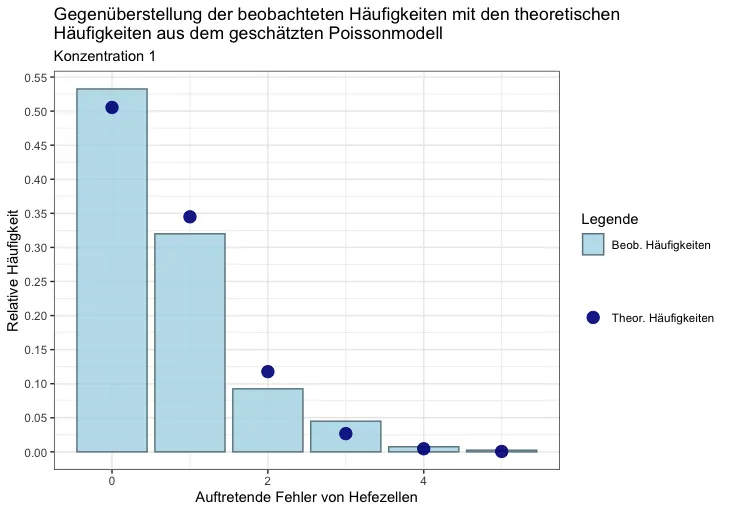 。
。
但是这个空隙很丑陋,应该有一种方法将这两个图例合并成一个,在其中两个条目彼此接近。 我已经花了超过9个小时来研究它,并查找了许多帖子,没有解决我的问题。 如果有任何不清楚的地方,请告诉我。正如我已经写过的,这是第一次提出问题。谢谢