我有两个3D点的.csv文件(数值坐标数据)和相关属性数据(字符串+数字)。我需要计算每个点与其他每个点之间的欧几里得距离,并保留每个点的属性数据,与差异相关联。我有一种方法可以实现此目的,但它使用了循环,我希望有更好的方法来完成这个任务,使其资源消耗更少。以下是我当前正在使用的代码:
import pandas as pd
import numpy as np
# read .csv
dataset_1 = pd.read_csv(dataset1 path)
dataset_2 = pd.read_csv(dataset2 path)
# convert to numpy array
array_1 = dataset_1.to_numpy()
array_2 = dataset_2.to_numpy()
# define data types for new array. This includes the attribute data I want to maintain
data_type = np.dtype('f4, f4, f4, U10, U10, f4, f4, f4, U10, U10, U10, f4, f4, U10, U100')
#define the new array
new_array = np.empty((len(array_1)*len(array_2)), dtype=data_type)
#calculate the Euclidean distance between each set of 3D coordinates, and populate the new array with the results as well as data from the input arrays
number3 = 0
for number in range(len(array_1)):
for number2 in range(len(array_2)):
Euclidean_Dist = np.linalg.norm(array_1[number, 0:3]-array_2[number2, 0:3])
new_array[number3] = (array_1[number, 0], array_1[number, 1], array_1[number, 2], array_1[number, 3], array_1[number, 7],
array_2[number2, 0], array_2[number2, 1],array_2[number2, 2], array_2[number2, 3], array_2[number2, 6], array_2[number2, 7],
array_2[number2, 12], array_2[number2, 13], dist,''.join(sorted((str(array_2[number2, 0]) + str(array_2[number2, 1]) + str(array_2[number2, 2]) + str(array_2[number2, 3])))))
number3+=1
#Convert results to pandas dataframe
new_df = pd.DataFrame(new_array)
我处理大型数据集,如果有人可以建议更有效的方法来完成这项工作,我将不胜感激。
谢谢,
上面介绍的代码适用于我的问题,但我正在寻找改进效率的方法。
编辑以显示示例输入数据集(dataset_1和dataset_2)和期望的输出数据集(new_df)。关键是对于输出数据集,我需要保留与欧几里德距离相关联的输入数据集的属性。我可以使用scipy.spatial.distance.cdist计算距离,但我不确定在输出数据中保留输入数据的属性的最佳方法。
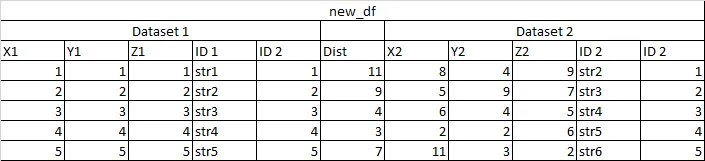
dataset_1和new_df的前五行吗?谢谢。 - Khaled DELLALdata_type不够高效。结构体数组(AoS)因其某些字段较大且其他字段可以受益于矢量化而被认为是低效的。请参见此帖子。这在这里尤其重要,因为Numpy没有有效地实现结构化类型。除此之外,您使用Unicode字符串,这也被认为是计算速度较慢的。如果您知道它们包含ASCII字符,请考虑使用ASCII字符串。还请注意,Numpy预留了160个32位字符的字符串空间,因此为640*X*Y字节。 - Jérôme RichardO(n log n)复杂度而不是O(n²)。所有这些都可以为足够大的数据(假设向量化正确执行)带来更快的代码执行速度。 - Jérôme Richard