设置
我目前正在尝试使用Sobel滤波器计算图像梯度。
起初,我使用了scipy.ndimage.sobel函数,
sx = ndimage.sobel(im, axis=0,mode="constant")
sy = ndimage.sobel(im, axis=1,mode="constant")
sobel = np.hypot(sx,sy)
sobel *= 255 / np.max(sobel)
然而,这仅适用于我的图像中的(3x3) Sobel滤波器,但我想尝试更大的滤波器。因此,我尝试使用numpy和scipy.signal计算图像梯度。首先,我再次尝试了(3x3)滤波器。
filter_x = np.array([[-1,0,1],[-2,0,2],[-1,0,1]], dtype=np.float)
filter_y = np.array([[1,2,1], [0,0,0], [-1,-2,-1]], dtype = np.float)
sx = signal.convolve2d(im,filter_x,mode="same",boundary="symm", fillvalue=0)
sy = signal.convolve2d(im,filter_y,mode="same",boundary="symm", fillvalue=0)
sobel = np.hypot(sx,sy)
sobel *= 255 / np.max(sobel)
如此帖中所建议的。
问题
不幸的是,这两种方法会导致完全不同的结果,这已经在此问题中提到过。
因此,我深入挖掘后发现scipy.ndimage.sobel使用的是correlate1d函数,而不是convolve2d或类似的任何东西(源代码)。不幸的是,由于其功能已被隐藏在已编译的_nd_image.pyd文件中,并位于我的conda环境中的site-packages文件夹中,因此无法查看correlate1d函数的内部源代码。因此,这里出现了我的问题:
问题
有人明确知道correlate1d正在计算什么以及它如何与convolve2d相比吗?
编辑
如Florian Drawitsch的答案中已经提到的,人们应该能够通过相关性替换卷积。但是,这些不同的结果是如何出现的?!
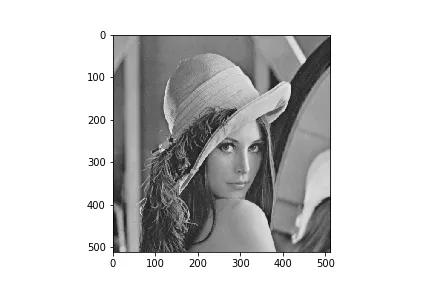

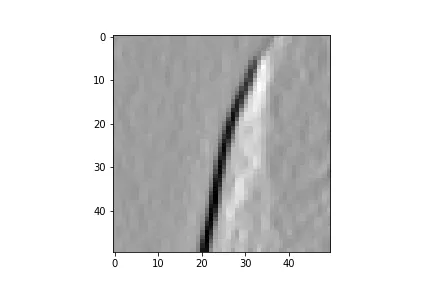


NI_Correlate1D函数位于同一文件夹中的 ni_filters.c 文件中。 - Reti43