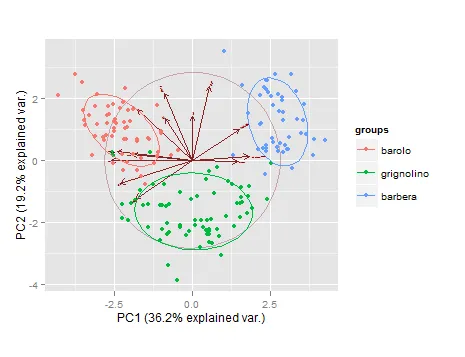
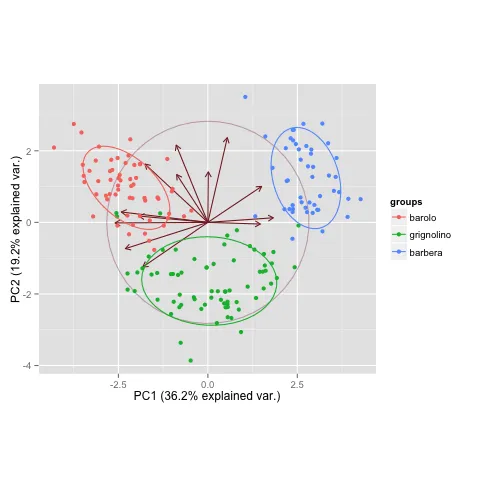
我有一个数据集data$cell_line.sva,其维度为313x11875。
cc.pca <- prcomp(data$cell_line.sva, center = TRUE, scale. = TRUE, retx = TRUE)
g <- ggbiplot(cc.pca, obs.scale = 1, var.scale = 1, groups = as.factor(cgpResponse), ellipse = TRUE, circle = FALSE)
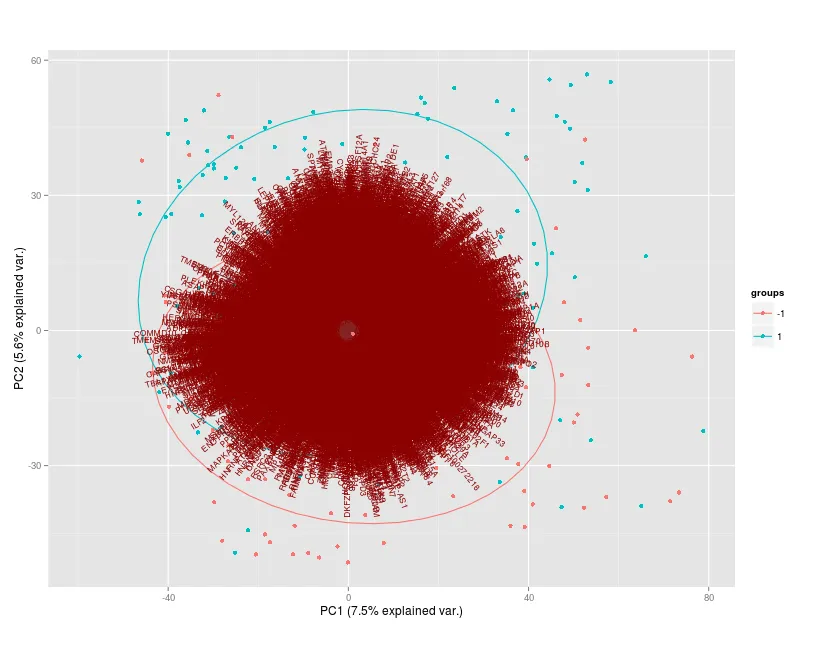
如何去掉特征名称?(红色文本)