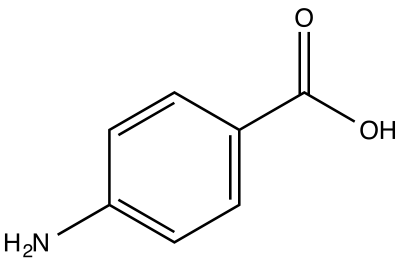
我希望在我的R绘图中添加4-氨基苯甲酸的分子结构。该绘图显示所述分子的红外光谱。是否可以使用SMILES代码添加,其为O=C(O)c1ccc(N)cc1,或者可以将其作为图片添加,可在此处找到(在底部进行了编辑)。我已编写以下脚本:
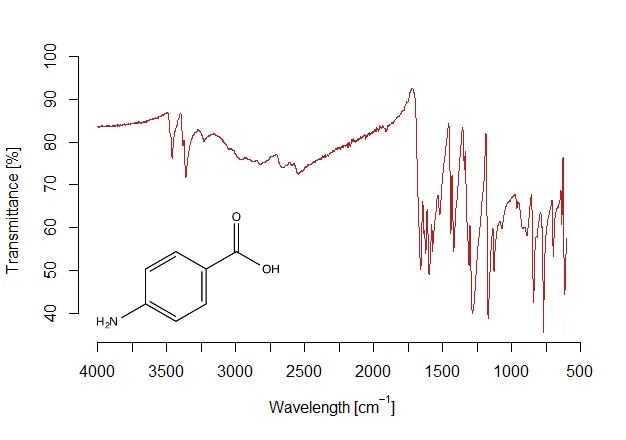
.asc文件可以在这里找到。 由于底部左侧空间最大,我希望分子能够被放置在那里。 图像缩小了85%:
par(family="mono", font.axis=1)
data <- read.table("D13-4-aminobenzoic_acid.asc")
x <- data[,1]
y <- data[,2]
x1 <- x[rev(order(x))] # reverse order x
plot(x1,y, type="n", xlim=rev(range(x)),
axes=FALSE,
xlab=expression(paste("Wavelength [", cm^-1,"]")),
ylab="Transmittance [%]"
)
lines(x1, y, col="firebrick")
axis(1, at=seq(500,4000,250))
axis(2, at=seq(40,100,10), xpd=T)
.asc文件可以在这里找到。 由于底部左侧空间最大,我希望分子能够被放置在那里。 图像缩小了85%:
image()命令。在我的研究中,我遇到了add.image()命令,它似乎类似于第一个提到的命令。然而,我以前从未在R中处理过图像,对此一无所知。 - user3182395