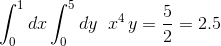可以通过以下方式在2D中完成。简要绘制一个点阵网格,
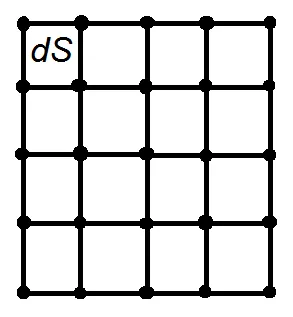
整个网格的积分等于小区域dS的积分之和。梯形法则将小矩形dS上的积分近似为dS的面积乘以dS角落处函数值的平均值,这些角落是网格点:
∫ f(x,y) dS = (f1 + f2 + f3 + f4)/4
其中f1、f2、f3、f4是矩形dS的角落处的数组值。
注意,每个内部网格点在整个积分公式中出现四次,因为它通常出现在四个矩形中。每个不在角落的边上的点出现两次,因为它通常出现在两个矩形中,而每个角点只出现一次。因此,numpy通过以下函数计算积分:
def double_Integral(xmin, xmax, ymin, ymax, nx, ny, A):
dS = ((xmax-xmin)/(nx-1)) * ((ymax-ymin)/(ny-1))
A_Internal = A[1:-1, 1:-1]
(A_u, A_d, A_l, A_r) = (A[0, 1:-1], A[-1, 1:-1], A[1:-1, 0], A[1:-1, -1])
(A_ul, A_ur, A_dl, A_dr) = (A[0, 0], A[0, -1], A[-1, 0], A[-1, -1])
return dS * (np.sum(A_Internal)\
+ 0.5 * (np.sum(A_u) + np.sum(A_d) + np.sum(A_l) + np.sum(A_r))\
+ 0.25 * (A_ul + A_ur + A_dl + A_dr))
在David GG提供的函数上进行测试:
x_min,x_max,n_points_x = (0,1,50)
y_min,y_max,n_points_y = (0,5,50)
x = np.linspace(x_min,x_max,n_points_x)
y = np.linspace(y_min,y_max,n_points_y)
def F(x,y):
return x**4 * y
zz = F(x.reshape(-1,1),y.reshape(1,-1))
print(double_Integral(x_min, x_max, y_min, y_max, n_points_x, n_points_y, zz))
2.5017353157550444
其他方法(Simpson,Romberg等)可以类似地推导出来。