另一种方法是生成自己的预测值,并使用ggplot绘制它们 - 这样你可以更好地控制最终的绘图(而不是依赖于stat_smooth进行计算;如果你使用多个协变量并需要在绘图时将某些常数保持在它们的均值或模式处,这尤其有用)。
library(ggplot2)
mydata <- data.frame(Ft = c(1, 6, 11, 16, 21, 2, 7, 12, 17, 22, 3, 8,
13, 18, 23, 4, 9, 14, 19, 5, 10, 15, 20),
Temp = c(66, 72, 70, 75, 75, 70, 73, 78, 70, 76, 69, 70,
67, 81, 58, 68, 57, 53, 76, 67, 63, 67, 79),
TD = c(0, 0, 1, 0, 1, 1, 0, 0, 0, 0, 0, 0,
0, 0, 1, 0, 1, 1, 0, 0, 1, 0, 0))
model <- glm(TD ~ Temp, data=mydata, family=binomial(link="logit"))
temp.data <- data.frame(Temp = seq(53, 81, 0.5))
predicted.data <- as.data.frame(predict(model, newdata = temp.data,
type="link", se=TRUE))
new.data <- cbind(temp.data, predicted.data)
std <- qnorm(0.95 / 2 + 0.5)
new.data$ymin <- model$family$linkinv(new.data$fit - std * new.data$se)
new.data$ymax <- model$family$linkinv(new.data$fit + std * new.data$se)
new.data$fit <- model$family$linkinv(new.data$fit)
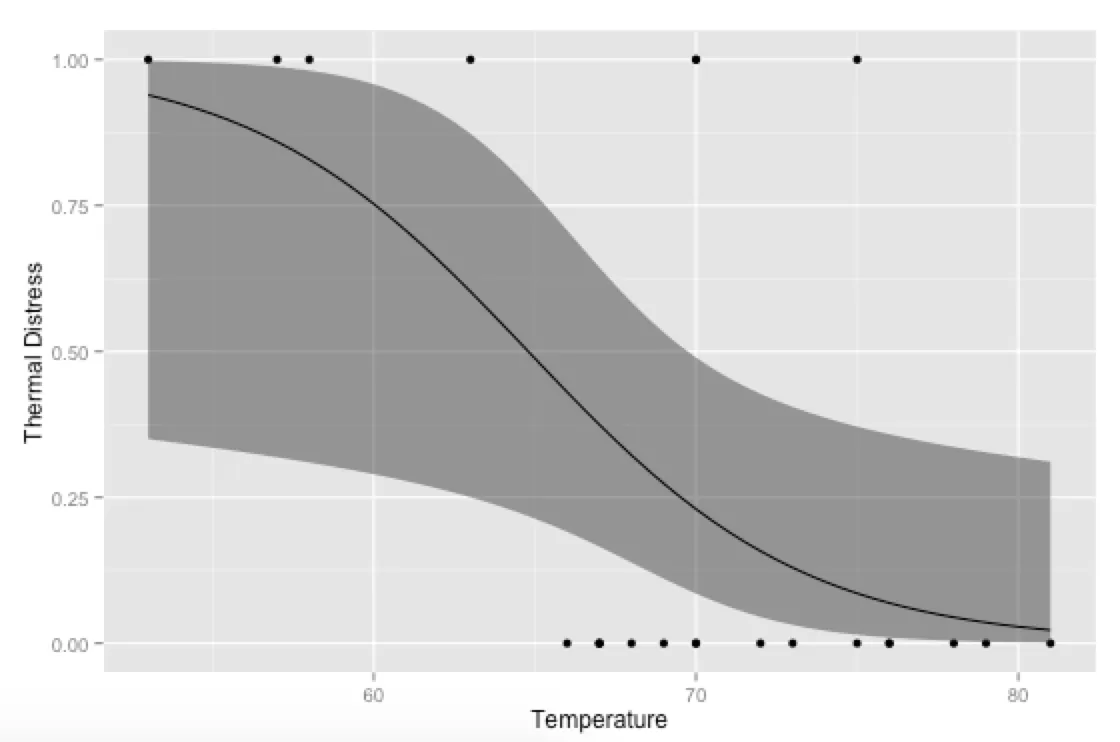
p <- ggplot(mydata, aes(x=Temp, y=TD))
p + geom_point() +
geom_ribbon(data=new.data, aes(y=fit, ymin=ymin, ymax=ymax), alpha=0.5) +
geom_line(data=new.data, aes(y=fit)) +
labs(x="Temperature", y="Thermal Distress")
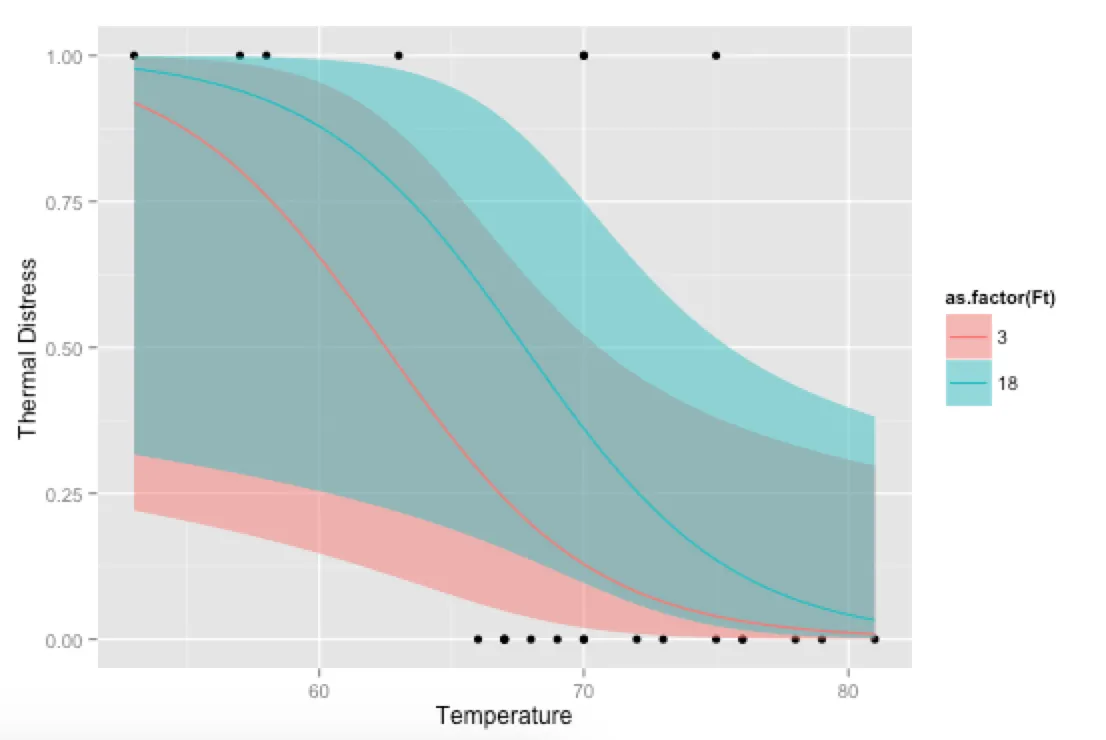
额外福利,只是为了好玩:如果您使用自己的预测函数,可以在协变量方面发疯,例如显示模型在不同水平的Ft下的拟合情况:
model <- glm(TD ~ Temp + Ft, data=mydata, family=binomial(link="logit"))
temp.data <- data.frame(Temp = rep(seq(53, 81, 0.5), 2),
Ft = c(rep(3, 57), rep(18, 57)))
predicted.data <- as.data.frame(predict(model, newdata = temp.data,
type="link", se=TRUE))
new.data <- cbind(temp.data, predicted.data)
std <- qnorm(0.95 / 2 + 0.5)
new.data$ymin <- model$family$linkinv(new.data$fit - std * new.data$se)
new.data$ymax <- model$family$linkinv(new.data$fit + std * new.data$se)
new.data$fit <- model$family$linkinv(new.data$fit)
p <- ggplot(mydata, aes(x=Temp, y=TD))
p + geom_point() +
geom_ribbon(data=new.data, aes(y=fit, ymin=ymin, ymax=ymax,
fill=as.factor(Ft)), alpha=0.5) +
geom_line(data=new.data, aes(y=fit, colour=as.factor(Ft))) +
labs(x="Temperature", y="Thermal Distress")


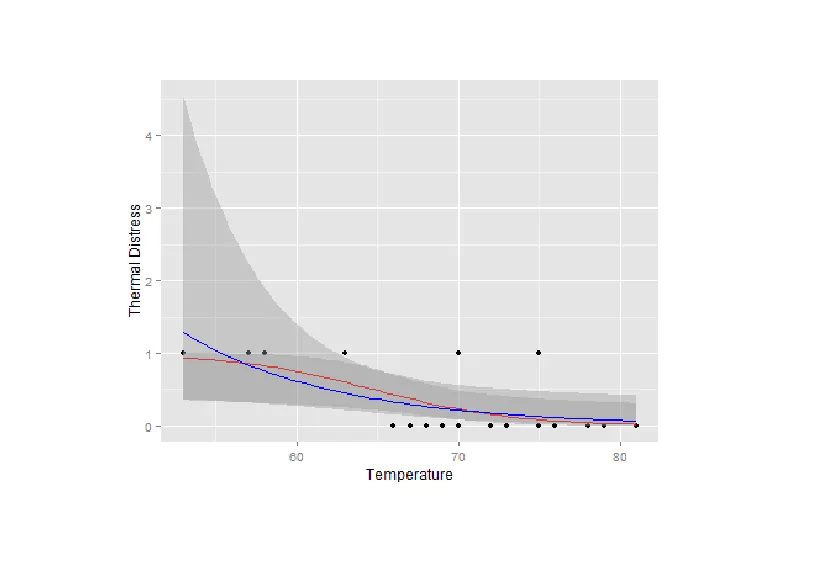
glm,你得到的置信区间超出了(0,1)的范围,这可能不是OP想要的... - Ben Bolkerdplyr可以大大简化所有数据框的创建过程,但是为了回答这个问题,我仍然坚持使用基本的R语言。 - Andrewscale_y_continuous()来定义 y 轴上的断点和标签,例如:scale_y_continuous(breaks=c(0, 1), labels=c("否", "是"))。 - Andrewpredict中使用参数type="response"是否有更简单的方法来获取conf.int(而不是调用model$family$linkinv)? - Marc in the box