我之前跳过了这个问题并使用了
davids answer(感谢!)。然而,后来事实证明,对于一个带有NaN的数组应用高斯滤波器的任务并不像我想象的那样明确定义。
正如
ndimage.gaussian_filter所描述的那样,在图像边界处理值时有不同的选项(反射,常数外推等)。类似的决定也必须针对图像中的NaN值做出。
- 有些想法可能是线性插值nan值,但问题是,如何处理图像边界处的nan值。
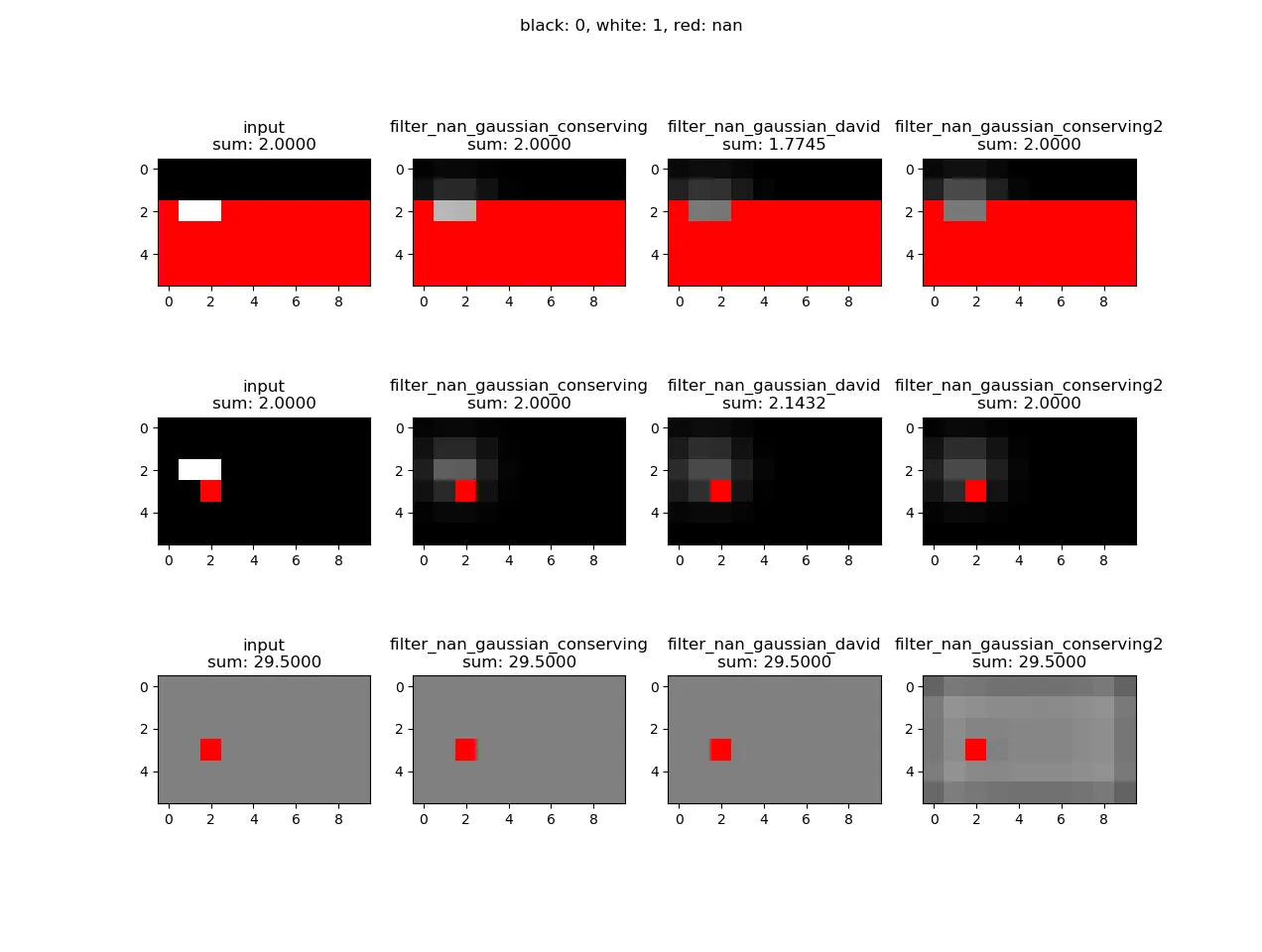
filter_nan_gaussian_david:David的方法等同于假设每个nan点都有一些平均邻域值。这会导致总强度的变化(请参见第3列中的sum值),但在其他方面表现出色。filter_nan_gaussian_conserving:此方法是通过高斯滤波器扩展每个点的强度。映射到nan像素的强度将被重新调整回原点。这是否有意义取决于应用程序。我有一个由nan包围的封闭区域,希望保留总强度+避免边界失真。filter_nan_gaussian_conserving2:通过高斯滤波器扩展每个点的强度。映射到nan像素的强度被重定向到具有相同高斯加权的其他像素。这导致在许多nan /边界像素附近的原点处强度的相对降低。这在最后一行的最右侧有说明。
代码
import numpy as np
from scipy import ndimage
import matplotlib as mpl
import matplotlib.pyplot as plt
def filter_nan_gaussian_conserving(arr, sigma):
"""Apply a gaussian filter to an array with nans.
Intensity is only shifted between not-nan pixels and is hence conserved.
The intensity redistribution with respect to each single point
is done by the weights of available pixels according
to a gaussian distribution.
All nans in arr, stay nans in gauss.
"""
nan_msk = np.isnan(arr)
loss = np.zeros(arr.shape)
loss[nan_msk] = 1
loss = ndimage.gaussian_filter(
loss, sigma=sigma, mode='constant', cval=1)
gauss = arr.copy()
gauss[nan_msk] = 0
gauss = ndimage.gaussian_filter(
gauss, sigma=sigma, mode='constant', cval=0)
gauss[nan_msk] = np.nan
gauss += loss * arr
return gauss
def filter_nan_gaussian_conserving2(arr, sigma):
"""Apply a gaussian filter to an array with nans.
Intensity is only shifted between not-nan pixels and is hence conserved.
The intensity redistribution with respect to each single point
is done by the weights of available pixels according
to a gaussian distribution.
All nans in arr, stay nans in gauss.
"""
nan_msk = np.isnan(arr)
loss = np.zeros(arr.shape)
loss[nan_msk] = 1
loss = ndimage.gaussian_filter(
loss, sigma=sigma, mode='constant', cval=1)
gauss = arr / (1-loss)
gauss[nan_msk] = 0
gauss = ndimage.gaussian_filter(
gauss, sigma=sigma, mode='constant', cval=0)
gauss[nan_msk] = np.nan
return gauss
def filter_nan_gaussian_david(arr, sigma):
"""Allows intensity to leak into the nan area.
According to Davids answer:
https://dev59.com/AGMl5IYBdhLWcg3wFjoC#36307291
"""
gauss = arr.copy()
gauss[np.isnan(gauss)] = 0
gauss = ndimage.gaussian_filter(
gauss, sigma=sigma, mode='constant', cval=0)
norm = np.ones(shape=arr.shape)
norm[np.isnan(arr)] = 0
norm = ndimage.gaussian_filter(
norm, sigma=sigma, mode='constant', cval=0)
norm = np.where(norm==0, 1, norm)
gauss = gauss/norm
gauss[np.isnan(arr)] = np.nan
return gauss
fig, axs = plt.subplots(3, 4)
fig.suptitle('black: 0, white: 1, red: nan')
cmap = mpl.cm.get_cmap('gist_yarg_r')
cmap.set_bad('r')
def plot_info(ax, arr, col):
kws = dict(cmap=cmap, vmin=0, vmax=1)
if col == 0:
title = 'input'
elif col == 1:
title = 'filter_nan_gaussian_conserving'
elif col == 2:
title = 'filter_nan_gaussian_david'
elif col == 3:
title = 'filter_nan_gaussian_conserving2'
ax.set_title(title + '\nsum: {:.4f}'.format(np.nansum(arr)))
ax.imshow(arr, **kws)
sigma = (1,1)
arr0 = np.zeros(shape=(6, 10))
arr0[2:, :] = np.nan
arr0[2, 1:3] = 1
arr1 = np.zeros(shape=(6, 10))
arr1[2, 1:3] = 1
arr1[3, 2] = np.nan
arr2 = np.ones(shape=(6, 10)) *.5
arr2[3, 2] = np.nan
plot_info(axs[0, 0], arr0, 0)
plot_info(axs[0, 1], filter_nan_gaussian_conserving(arr0, sigma), 1)
plot_info(axs[0, 2], filter_nan_gaussian_david(arr0, sigma), 2)
plot_info(axs[0, 3], filter_nan_gaussian_conserving2(arr0, sigma), 3)
plot_info(axs[1, 0], arr1, 0)
plot_info(axs[1, 1], filter_nan_gaussian_conserving(arr1, sigma), 1)
plot_info(axs[1, 2], filter_nan_gaussian_david(arr1, sigma), 2)
plot_info(axs[1, 3], filter_nan_gaussian_conserving2(arr1, sigma), 3)
plot_info(axs[2, 0], arr2, 0)
plot_info(axs[2, 1], filter_nan_gaussian_conserving(arr2, sigma), 1)
plot_info(axs[2, 2], filter_nan_gaussian_david(arr2, sigma), 2)
plot_info(axs[2, 3], filter_nan_gaussian_conserving2(arr2, sigma), 3)
plt.show()
