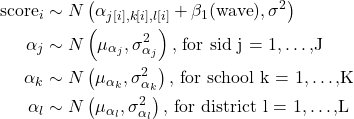{equatiomatic}的小插图包括以下示例(此处):
这导致了如下的符号表示:
当然,这个模型对于虚构的数据完全不适用,但是暂且不考虑这一点。{equatiomatic} 的输出如下: 与小品例子中一样,我们有多个随机截距和一个固定斜率。但是方程看起来非常不同。斜率已经移动到第三行,进入了
我真的不明白符号差异的原因,或者说,为什么斜率要进入
library(lme4)
library(equatiomatic)
lev1_long <- lmer(score ~ wave + (1|sid) + (1|school) + (1|district),
data = sim_longitudinal)
extract_eq(lev1_long)
这导致了如下的符号表示:
不同的随机截距在各自的行中被标记出来(我认为暗示它们相加以生成组合截距?),并且固定斜率wave,表示为 $\beta_1$,被包含在第一行中,在结果变量 score 的分布中。我可以成功地复制这个例子。
然而,当我使用一些数据进行操作时,我得到了不同的符号。这里有一个用虚拟数据集可重复的示例:
library(tidyverse)
library(lme4)
library(equatiomatic)
mock_df <- tibble(
outcome = 1:10,
oID = c('D', 'A', 'B', 'C', 'B', 'B', 'E', 'B', 'A', 'C'),
treatment = rep(c(0, 1), each = 5),
pID = c('P1', 'P1', 'P2', 'P2', 'P2', 'P3', 'P3', 'P3', 'P4', 'P4'))
model <- lmer(outcome ~ 1 + treatment + (1|pID) + (1|oID), data = mock_df)
extract_eq(model)
当然,这个模型对于虚构的数据完全不适用,但是暂且不考虑这一点。{equatiomatic} 的输出如下: 与小品例子中一样,我们有多个随机截距和一个固定斜率。但是方程看起来非常不同。斜率已经移动到第三行,进入了
pID 的随机截距分布中。我真的不明白符号差异的原因,或者说,为什么斜率要进入
pID 截距的分布而不是 oID 截距的分布。在示例中和我制作的虚构模拟示例之间是否存在实际的基本模型结构差异,这是否可以证明符号差异的合理性?