我正在使用NumPy学习图像处理,并在用卷积进行滤波时遇到问题。
我想对灰度图像进行卷积。 (将一个二维数组与一个较小的二维数组进行卷积)
有没有人有办法改进我的方法?
我知道SciPy支持convolve2d,但我想只使用NumPy来实现卷积。
我已经做了什么
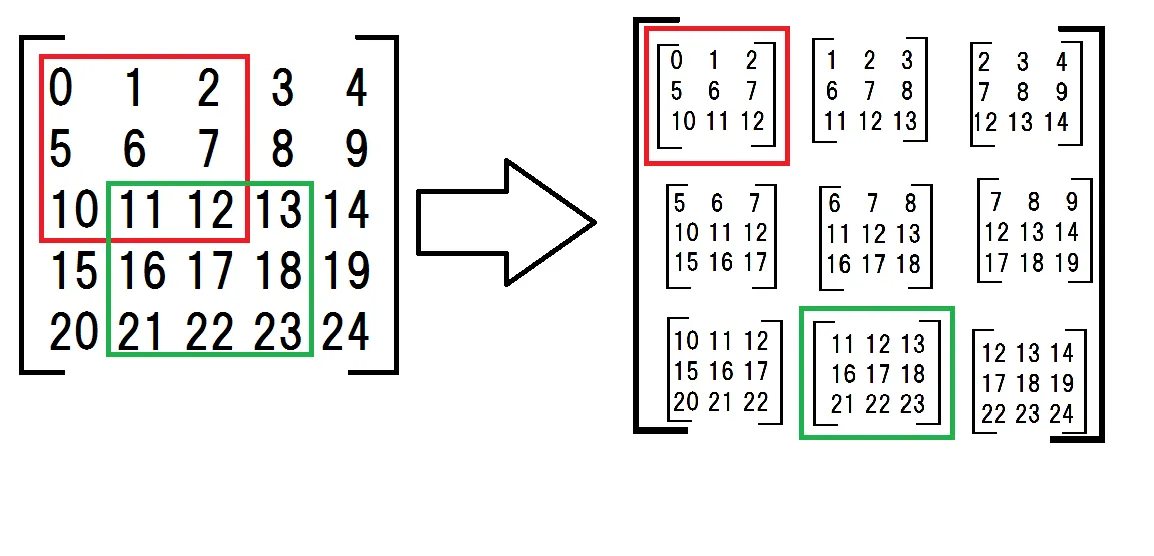
首先,我制作了一个由子矩阵组成的二维数组。
a = np.arange(25).reshape(5,5) # original matrix
submatrices = np.array([
[a[:-2,:-2], a[:-2,1:-1], a[:-2,2:]],
[a[1:-1,:-2], a[1:-1,1:-1], a[1:-1,2:]],
[a[2:,:-2], a[2:,1:-1], a[2:,2:]]])
子矩阵看起来很复杂,但我正在做的内容如下图所示。
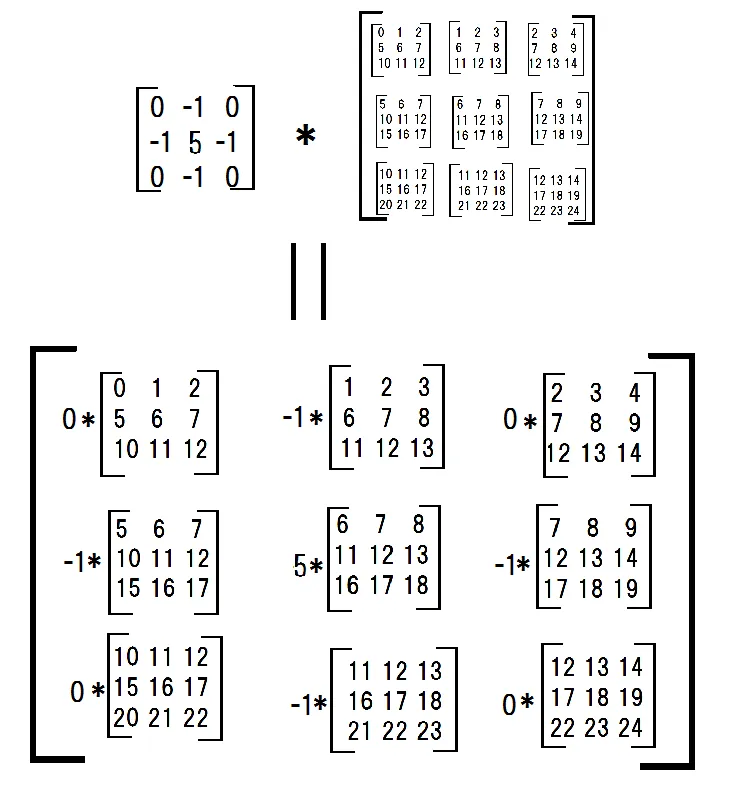
接下来,我将每个子矩阵与一个滤波器相乘。
conv_filter = np.array([[0,-1,0],[-1,4,-1],[0,-1,0]])
multiplied_subs = np.einsum('ij,ijkl->ijkl',conv_filter,submatrices)
并将它们相加。
np.sum(np.sum(multiplied_subs, axis = -3), axis = -3)
#array([[ 6, 7, 8],
# [11, 12, 13],
# [16, 17, 18]])
因此,这个过程可以被称为我的convolve2d。
def my_convolve2d(a, conv_filter):
submatrices = np.array([
[a[:-2,:-2], a[:-2,1:-1], a[:-2,2:]],
[a[1:-1,:-2], a[1:-1,1:-1], a[1:-1,2:]],
[a[2:,:-2], a[2:,1:-1], a[2:,2:]]])
multiplied_subs = np.einsum('ij,ijkl->ijkl',conv_filter,submatrices)
return np.sum(np.sum(multiplied_subs, axis = -3), axis = -3)
然而,我发现我的my_convolve2d有3个令人困扰的问题:
- 生成子矩阵太棘手了,难以阅读,只能在过滤器为3*3时使用。
- 变量子矩阵的大小似乎太大了,因为它大约比原始矩阵大9倍。
- 求和似乎有点不直观。简单来说,很丑陋。
感谢您阅读到这里。
更新一下,我为自己编写了一个conv3d。我将把它留作公共领域。
def convolve3d(img, kernel):
# calc the size of the array of submatrices
sub_shape = tuple(np.subtract(img.shape, kernel.shape) + 1)
# alias for the function
strd = np.lib.stride_tricks.as_strided
# make an array of submatrices
submatrices = strd(img,kernel.shape + sub_shape,img.strides * 2)
# sum the submatrices and kernel
convolved_matrix = np.einsum('hij,hijklm->klm', kernel, submatrices)
return convolved_matrix