我认为我们可以进行“有效”的FFT卷积,并仅选择跨步位置上的结果,如下所示:
def strideConv(arr,arr2,s):
cc=scipy.signal.fftconvolve(arr,arr2[::-1,::-1],mode='valid')
idx=(np.arange(0,cc.shape[1],s), np.arange(0,cc.shape[0],s))
xidx,yidx=np.meshgrid(*idx)
return cc[yidx,xidx]
这与其他人的答案给出了相同的结果。但我猜只有当内核大小为奇数时才有效。
另外,我在
arr2[::-1, ::-1]中翻转了内核,只是为了与其他人保持一致,根据上下文,您可能想省略它。
更新:
目前,我们有几种使用numpy和scipy进行2D或3D卷积的不同方法,我考虑进行一些比较,以便了解哪种方法在不同大小的数据上更快。希望这不会被视为离题。
方法1:FFT卷积(使用
scipy.signal.fftconvolve):
def padArray(var,pad,method=1):
if method==1:
var_pad=numpy.zeros(tuple(2*pad+numpy.array(var.shape[:2]))+var.shape[2:])
var_pad[pad:-pad,pad:-pad]=var
else:
var_pad=numpy.pad(var,([pad,pad],[pad,pad])+([0,0],)*(numpy.ndim(var)-2),
mode='constant',constant_values=0)
return var_pad
def conv3D(var,kernel,stride=1,pad=0,pad_method=1):
'''3D convolution using scipy.signal.convolve.
'''
var_ndim=numpy.ndim(var)
kernel_ndim=numpy.ndim(kernel)
stride=int(stride)
if var_ndim<2 or var_ndim>3 or kernel_ndim<2 or kernel_ndim>3:
raise Exception("<var> and <kernel> dimension should be in 2 or 3.")
if var_ndim==2 and kernel_ndim==3:
raise Exception("<kernel> dimension > <var>.")
if var_ndim==3 and kernel_ndim==2:
kernel=numpy.repeat(kernel[:,:,None],var.shape[2],axis=2)
if pad>0:
var_pad=padArray(var,pad,pad_method)
else:
var_pad=var
conv=fftconvolve(var_pad,kernel,mode='valid')
if stride>1:
conv=conv[::stride,::stride,...]
return conv
方法2:特殊转换(参见
此答案):
def conv3D2(var,kernel,stride=1,pad=0):
'''3D convolution by sub-matrix summing.
'''
var_ndim=numpy.ndim(var)
ny,nx=var.shape[:2]
ky,kx=kernel.shape[:2]
result=0
if pad>0:
var_pad=padArray(var,pad,1)
else:
var_pad=var
for ii in range(ky*kx):
yi,xi=divmod(ii,kx)
slabii=var_pad[yi:2*pad+ny-ky+yi+1:1, xi:2*pad+nx-kx+xi+1:1,...]*kernel[yi,xi]
if var_ndim==3:
slabii=slabii.sum(axis=-1)
result+=slabii
if stride>1:
result=result[::stride,::stride,...]
return result
第三种方法:Divakar建议的步幅视图卷积。
def asStride(arr,sub_shape,stride):
'''Get a strided sub-matrices view of an ndarray.
<arr>: ndarray of rank 2.
<sub_shape>: tuple of length 2, window size: (ny, nx).
<stride>: int, stride of windows.
Return <subs>: strided window view.
See also skimage.util.shape.view_as_windows()
'''
s0,s1=arr.strides[:2]
m1,n1=arr.shape[:2]
m2,n2=sub_shape[:2]
view_shape=(1+(m1-m2)//stride,1+(n1-n2)//stride,m2,n2)+arr.shape[2:]
strides=(stride*s0,stride*s1,s0,s1)+arr.strides[2:]
subs=numpy.lib.stride_tricks.as_strided(arr,view_shape,strides=strides)
return subs
def conv3D3(var,kernel,stride=1,pad=0):
'''3D convolution by strided view.
'''
var_ndim=numpy.ndim(var)
kernel_ndim=numpy.ndim(kernel)
if var_ndim<2 or var_ndim>3 or kernel_ndim<2 or kernel_ndim>3:
raise Exception("<var> and <kernel> dimension should be in 2 or 3.")
if var_ndim==2 and kernel_ndim==3:
raise Exception("<kernel> dimension > <var>.")
if var_ndim==3 and kernel_ndim==2:
kernel=numpy.repeat(kernel[:,:,None],var.shape[2],axis=2)
if pad>0:
var_pad=padArray(var,pad,1)
else:
var_pad=var
view=asStride(var_pad,kernel.shape,stride)
if numpy.ndim(kernel)==2:
conv=numpy.sum(view*kernel,axis=(2,3))
else:
conv=numpy.sum(view*kernel,axis=(2,3,4))
return conv
我进行了3组比较:
- 对2D数据进行卷积,使用不同的输入大小和卷积核大小,步长为1,填充为0。结果如下(颜色表示卷积重复10次所用的时间):
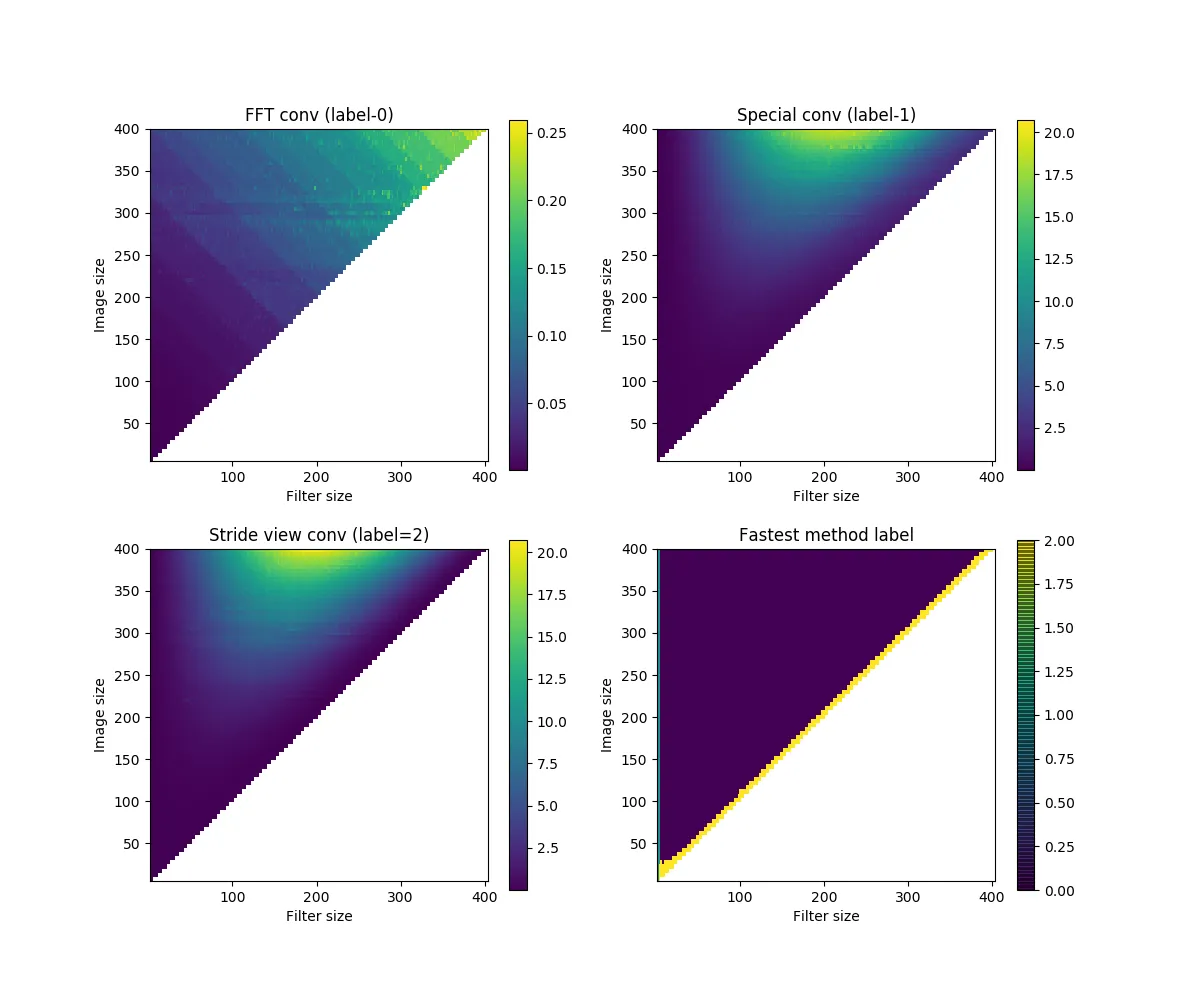
"FFT conv"通常是最快的。当卷积核大小增加时,“Special conv”和“Stride-view conv”变慢,但随着接近输入数据的大小,又会减少。最后一个子图显示了最快的方法,因此紫色的大三角形表示FFT是赢家,但请注意左侧有一条细绿色柱(可能太小看不清楚,但确实存在),这表明“Special conv”对于非常小的内核(小于约5x5)具有优势。而当内核大小接近输入时,“stride-view conv”是最快的(见对角线)。
比较2:对3D数据进行卷积。
设置:pad=0,stride=2,输入维度=
nxnx5,内核形状=
fxfx5。
当内核大小位于输入的中间时,我跳过了“Special Conv”和“Stride-view conv”的计算。基本上,“Special Conv”现在没有优势,“Stride-view”比FFT对于小和大内核都更快。
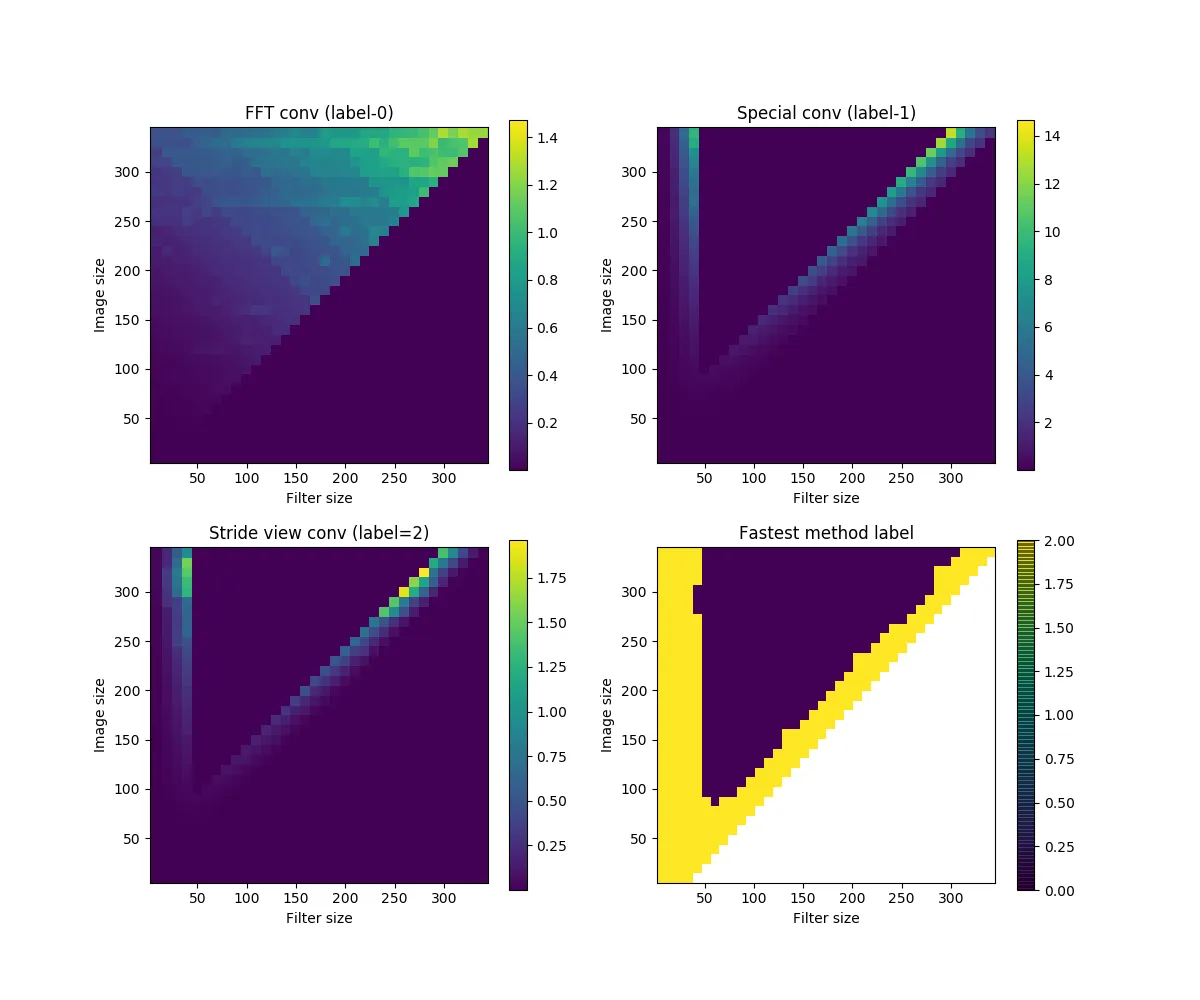
补充说明:当尺寸超过350时,我注意到“Stride-view conv”会出现相当大的内存使用峰值。
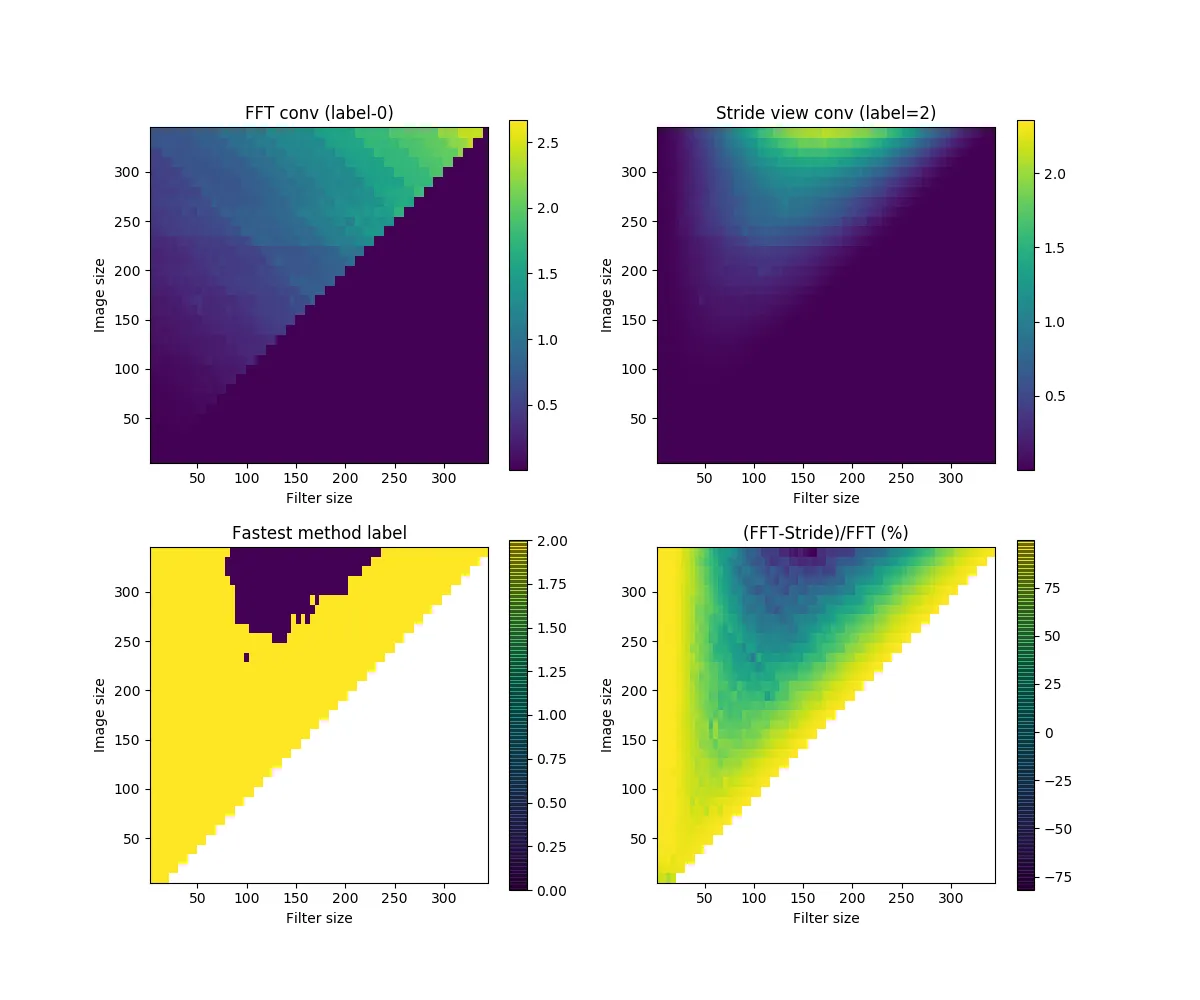
比较3:使用更大步长对3D数据进行卷积。
设置:填充=0,步长=5,输入维度=nxnx10,内核形状=fxfx10。
这次我省略了“特殊卷积”。对于更大的区域,“Stride-view conv”超越了FFT,最后一个子图显示差异接近100%。
可能是因为随着步幅增加,FFT方法将有更多的浪费数字,因此“stride-view”在小型和大型内核方面获得更多优势。



p没有被使用? - Divakar1x1x10x10的单个图像和一个大小为1x1x3x3的单个过滤器。...然后,天真地说,如果我们要对图像进行卷积操作,则会在图像上循环,并在每个位置上进行点积...”和“但是,如果我们不想执行循环怎么办?...我们需要收集可以应用过滤器的所有可能位置,然后进行单个矩阵乘法以获得每个可能位置的点积。” - dfrib