首先,您正在使用错误的方向输入数据。正如您可以从文档中看到的那样:
fit(PCA, X; ...)
对给定矩阵X执行PCA。X的每一列是一个观察值。
您需要将观察值放在列上,变量放在行上。尽管我们通常认为变量应该是列,但在基础线性代数的背景下,这样做更有意义。因此,为了正确处理数据,请从以下开始:
using MultivariateStats, Statistics
using RDatasets: dataset
iris = dataset("datasets", "iris")
iris_matrix = Array(iris[:, 1:4])'
M = fit(PCA, iris_matrix; pratio=1, maxoutdim=4)
正如您所注意到的,这将返回一个PCA类型的对象。事实证明,在文档中详细描述了此类型的可用方法:
属性
设M是PCA的实例,d为观察值的维度,p为输出维度(即主子空间的维度)
indim(M)
获取输入维数d,即观测空间的维数。
outdim(M)
获取输出维数p,即主子空间的维数。
mean(M)
获取均值向量(长度为d)。
projection(M)
获取投影矩阵(大小为(d, p))。投影矩阵的每列对应于一个主成分。
主成分按相应方差的降序排列。
principalvars(M)
主成分的方差。
tprincipalvar(M)
主成分的总方差,等于sum(principalvars(M))。
tresidualvar(M)
总剩余方差。
tvar(M)
观测总方差,等于tprincipalvar(M) + tresidualvar(M)。
principalratio(M)
主子空间中保留的方差比,等于tprincipalvar(M) / tvar(M)。
但是,如果您不知道这一点或者在找文档时遇到困难,您可以通过使用methodswith函数获取所有可用方法的完整列表(对于Julia中的任何类型)-- 在本例中特别是methodswith(PCA)。
其中最重要的是projection,它给出了实际的投影矩阵:
julia> proj = projection(M)
4×4 Matrix{Float64}:
-0.361387 0.656589 -0.58203 0.315487
0.0845225 0.730161 0.597911 -0.319723
-0.856671 -0.173373 0.0762361 -0.479839
-0.358289 -0.075481 0.545831 0.753657
根据文档所述,投影矩阵的每一列对应一个主成分。
因此,proj[:,1] 包含PC1的权重/“载荷”,proj[:,2] 包含PC2的载荷,以此类推。
由于您询问了贡献,我猜您是指每个主成分对解释总方差的贡献,文档告诉我们可以使用 principalvars 来获取它们:
julia> principalvars(M)
4-element Vector{Float64}:
4.2282417060348605
0.24267074792863352
0.07820950004291898
0.023835092973449976
因此,PC1在这里确实承担了大部分的重要任务。或者如果你更喜欢以每个成分解释的总方差的百分比来表达,则为:
julia> principalvars(M) ./ tvar(M) * 100
4-element Vector{Float64}:
92.46187232017269
5.306648311706788
1.7102609807929683
0.5212183873275495
预览同一文档的“转换”部分
转换和构造
给定PCA模型M,可以使用它将观测值转换为主成分,如下所示:
= ᵀ(-)
或使用它来从主成分(近似)重构观测数据,如下所示:
̃ = +
这里, 是投影矩阵。
该软件包提供了相应的方法:
transform(M, x)
将观测数据x转换为主成分。
其中,x可以是长度为d的向量或每列代表一个观测的矩阵。
reconstruct(M, y)
根据y中提供的主成分近似重构观测数据。
其中,y可以是长度为p的向量或每列代表某个观测的主成分的矩阵。
我们可以看到,将此转换应用于我们的数据的方法是
iris_transformed = transform(M, iris_matrix)
或者如果您想自己进行线性代数计算
iris_transformed = projection(M)' * (iris_matrix .- mean(M))
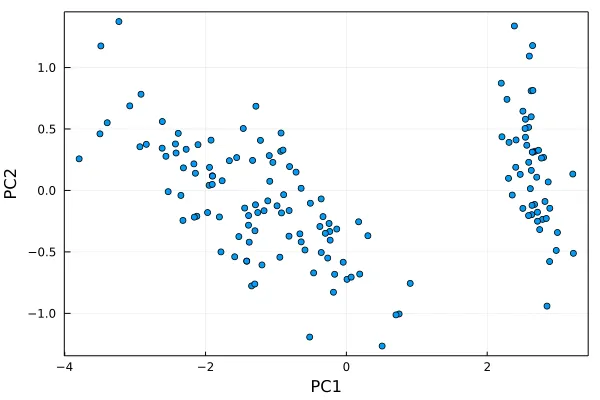
在获得转换后的数据之后,我们可以使用某种方法将前两个主成分绘制在一起,例如:
using Plots
h = plot(iris_transformed[1,:], iris_transformed[2,:], seriestype=:scatter, label="")
plot!(xlabel="PC1", ylabel="PC2", framestyle=:box)
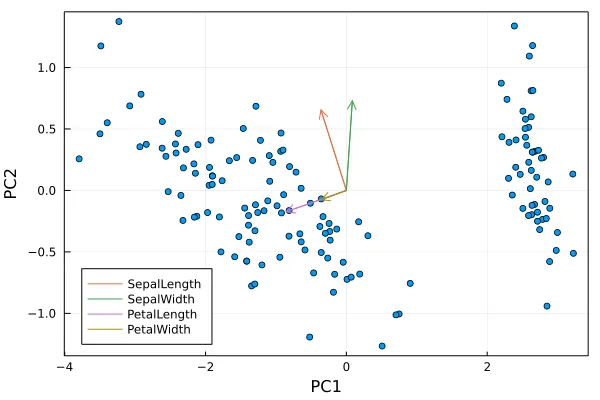
最后,如果您想为不同变量添加箭头,那么我们已经在projection(M)中拥有了所有需要的内容,而这已经被存储为proj
for i=1:4
display(h)
编辑:我应该也提一下,虽然目前没有StatsPlots适用于PCA类型的配方,但是有一个适用于MDS的方法,因此您可以通过简单地编写以下代码获得与上述相同的图:
M = fit(MDS, iris_matrix
using StatsPlots
plot(M)
(在这种情况下,当距离度量为原始(即高维)向量空间中的欧几里得距离时,PCA和MDS是等价的)
PCA和MDS在这种情况下是等价的,因为距离度量采用原始向量空间中的欧几里得距离。

