我正在尝试创建一个调整后的生存曲线(来自Cox模型),希望将此信息显示为累积事件。
我已经尝试过这个方法:
因为在
我刚接触R环境和ggplot,我该如何使用新创建的列绘制调整后的生存曲线,并保留原始图中的主题(包含在gplot中)?
谢谢!
编辑:
我的当前解决方案如下。
这达到了我想要的生存曲线,但我不确定置信度条是否真的是标准误差(+/-1)。我向
如何进一步自定义此图形,因为基础图形看起来相当单调!如何获得与survminer曲线尽可能接近的显示?
我已经尝试过这个方法:
library(survival)
data("ovarian")
library(survminer)
model<-coxph(Surv(futime, fustat) ~ age + strata(rx), data=ovarian)
gplot<-ggadjustedcurves(model) ## Expected plot of adjusted survival curve
因为在
ggadjustedcurves中还没有实现"fun=",所以我采纳了此页面上的用户的建议,将元素提取到plotdata中,并创建了一个新列,如下所示。plotdata<-gplot$data
plotdata%<>%
mutate(new=1-surv) ## 1-survival probability
我刚接触R环境和ggplot,我该如何使用新创建的列绘制调整后的生存曲线,并保留原始图中的主题(包含在gplot中)?
谢谢!
编辑:
我的当前解决方案如下。
library(rms)
model<-coxph(Surv(futime, fustat) ~ age+ strata(rx), data=ovarian)
survfit(model, conf.type = "plain", conf.int = 1)
plot(survfit(model), conf.int = T,col = c(1,2), fun='event')
这达到了我想要的生存曲线,但我不确定置信度条是否真的是标准误差(+/-1)。我向
conf.int参数提供了1,并相信以这种方式创建了标准误差,因为conf.type指定为plain。如何进一步自定义此图形,因为基础图形看起来相当单调!如何获得与survminer曲线尽可能接近的显示?
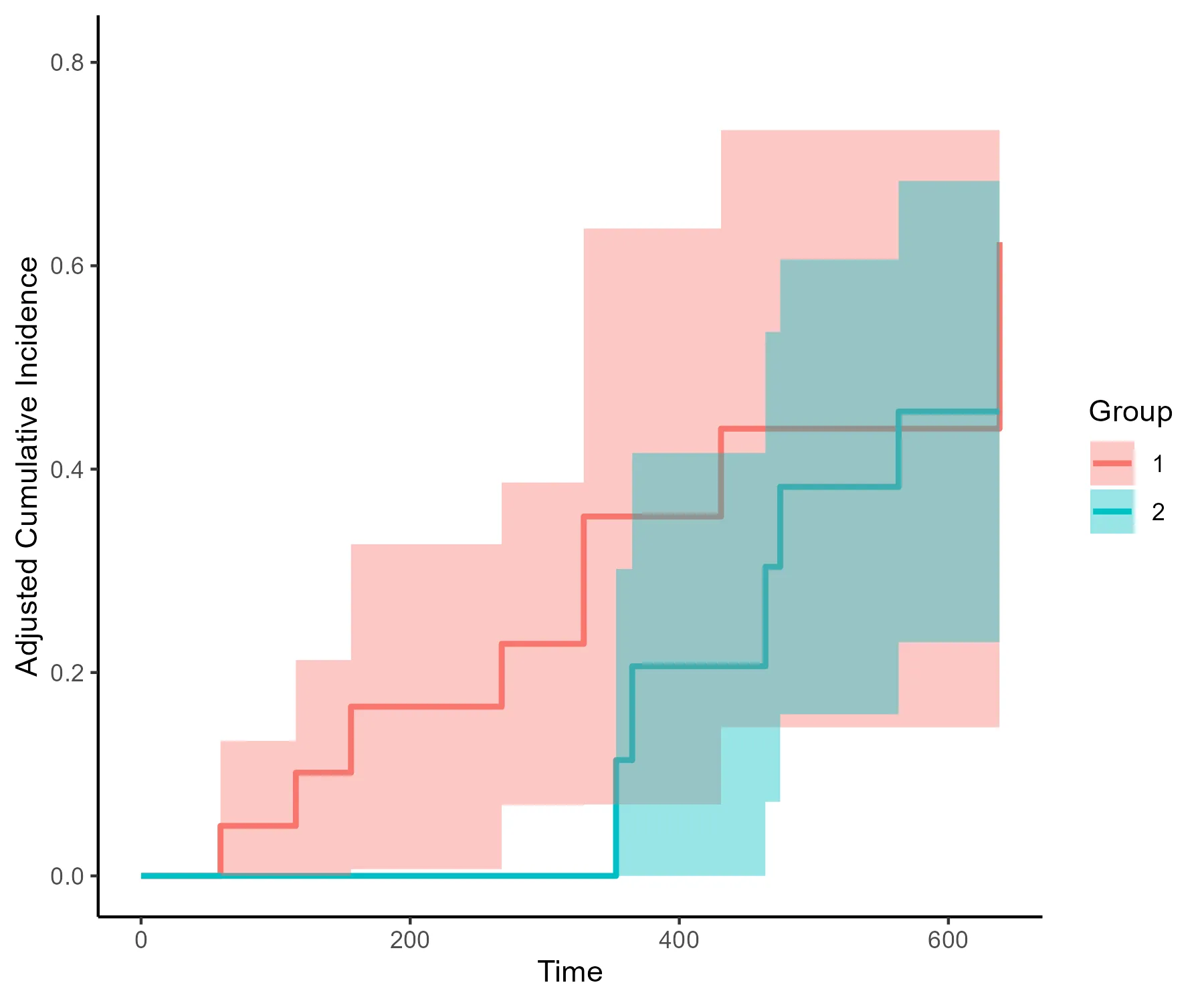
survfit和plot.survfit都来自survival包。(这些信息可能是必要的,也可能不是必要的,因为加载“rms”将可能加载“survival”,但它澄清了在哪里找到适当的帮助页面。)通常惯例是在1.96 *(+/-se)处绘制置信区间。 - IRTFM