有三种快速的方法来读取多个文件并将它们放入一个数据框或数据表中
首先获取所有txt文件的列表(包括子文件夹中的文件)
list_of_files <- list.files(path = ".", recursive = TRUE,
pattern = "\\.txt$",
full.names = TRUE)
1)使用fread()和rbindlist()函数来自data.table包
library(data.table)
DT <- rbindlist(sapply(list_of_files, fread, simplify = FALSE),
use.names = TRUE, idcol = "FileName")
2)使用readr :: read_table2()与purrr :: map_df()从tidyverse框架:
library(tidyverse)
df <- list_of_files %>%
set_names(.) %>%
map_df(read_table2, .id = "FileName")
3) (可能是三个中最快的)使用vroom::vroom():
library(vroom)
df <- vroom(list_of_files, .id = "FileName")
注意: 为了清理文件名,请使用basename或gsub函数。
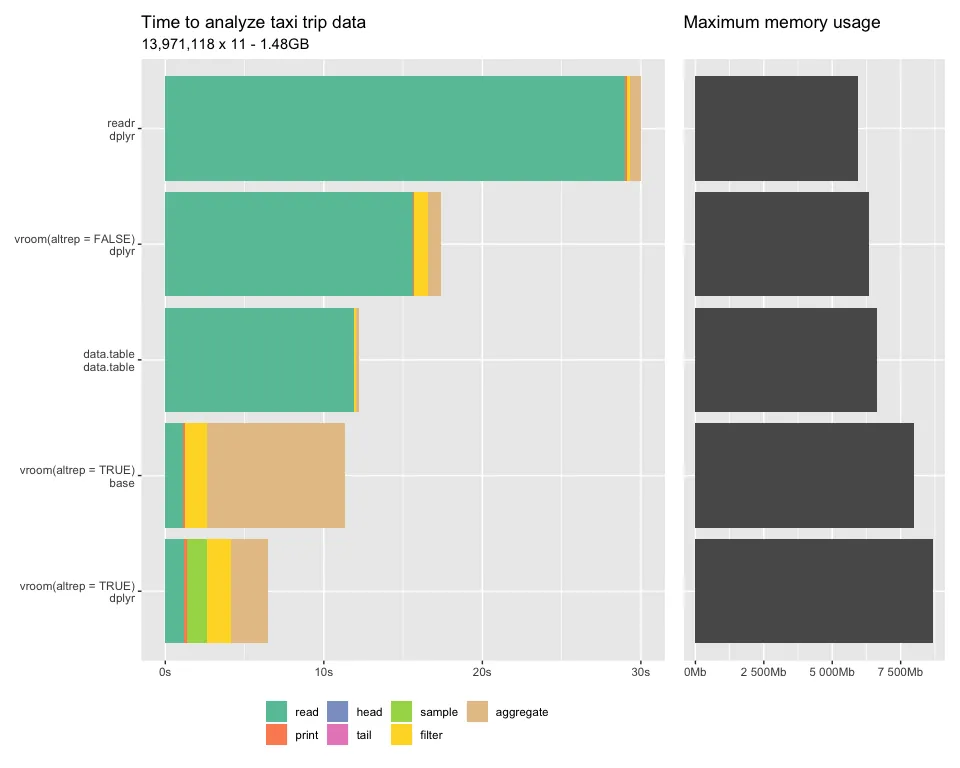
基准测试: readr与data.table和vroom在处理大数据方面的比较。
编辑1: 使用readr::read_csv读取多个csv文件并跳过header。
list_of_files <- list.files(path = ".", recursive = TRUE,
pattern = "\\.csv$",
full.names = TRUE)
df <- list_of_files %>%
purrr::set_names(nm = (basename(.) %>% tools::file_path_sans_ext())) %>%
purrr::map_df(read_csv,
col_names = FALSE,
skip = 1,
.id = "FileName")
编辑 2: 如果要将包含通配符的模式转换为等效的正则表达式,请使用glob2rx()

txt不是一个函数。你指向的链接是关于read.csv函数的。 - Wok