我正在使用sklearn的PCA对大量图像进行降维处理。一旦完成PCA拟合,我想查看组件的外观。
可以通过查看components_属性来实现这一点。由于不知道该属性的存在,我选择了其他方法:
each_component = np.eye(total_components)
component_im_array = pca.inverse_transform(each_component)
for i in range(num_components):
component_im = component_im_array[i, :].reshape(height, width)
# do something with component_im
换句话说,我在PCA空间中创建了一个图像,其中除1个特征外,所有特征均设为0。通过反向转换它们,然后应该得到原始空间中的图像,该图像一旦转换,就可以仅使用该PCA组件来表达。
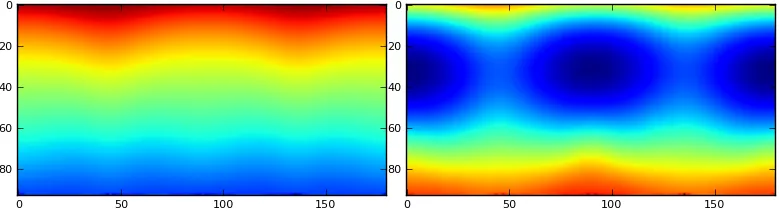
下图显示了结果。左边是使用我的方法计算得出的分量。右边是直接使用
pca.components_[i]。此外,使用我的方法,大多数图像非常相似(但它们确实是不同的),而通过访问components_,图像非常不同,正如我所预期的那样。我的方法是否存在概念问题?显然,从
pca.components_[i]获取的组件是正确的(或者至少更正确)。谢谢!
[0,0,...,0,1,0,...,0],这个图像必须来自一个看起来像组件的原始图像。 - Miquel