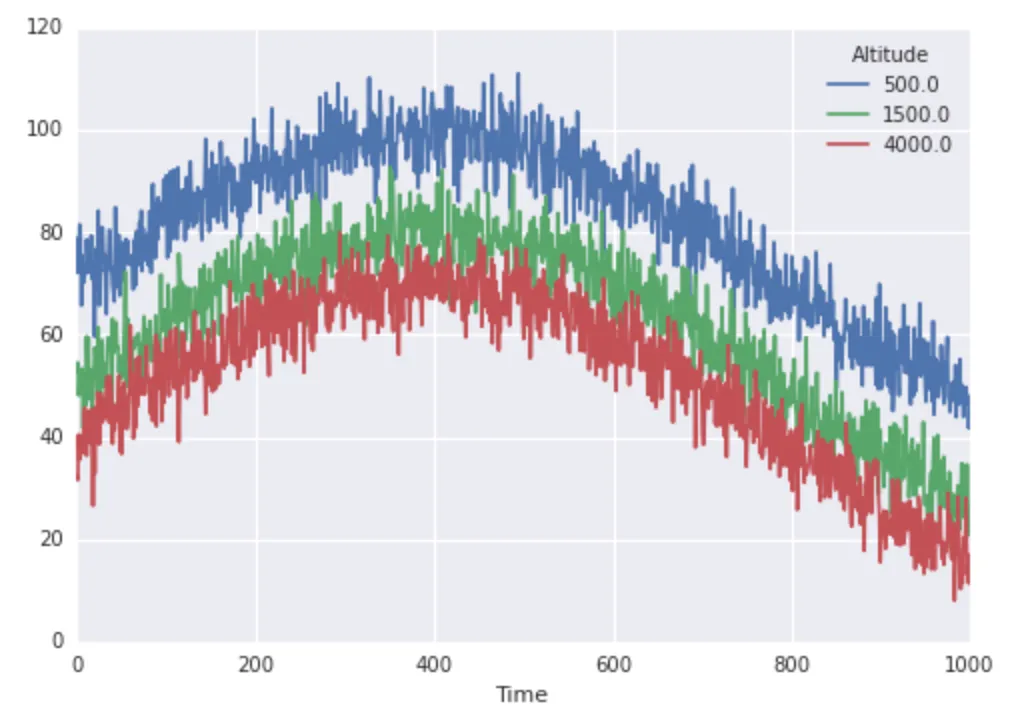
假设我有来自山上三个(已知)高度的气象站数据。具体而言,每个站每分钟记录其位置的温度测量值。我想执行两种插值,并希望能够快速执行每种插值。
因此,让我们设置一些数据:
import numpy as np
from scipy.interpolate import interp1d
import pandas as pd
import seaborn as sns
np.random.seed(0)
N, sigma = 1000., 5
basetemps = 70 + (np.random.randn(N) * sigma)
midtemps = 50 + (np.random.randn(N) * sigma)
toptemps = 40 + (np.random.randn(N) * sigma)
alltemps = np.array([basetemps, midtemps, toptemps]).T # note transpose!
trend = np.sin(4 / N * np.arange(N)) * 30
trend = trend[:, np.newaxis]
altitudes = np.array([500, 1500, 4000]).astype(float)
finaltemps = pd.DataFrame(alltemps + trend, columns=altitudes)
finaltemps.index.names, finaltemps.columns.names = ['Time'], ['Altitude']
finaltemps.plot()
将所有时间插值到相同的高度:
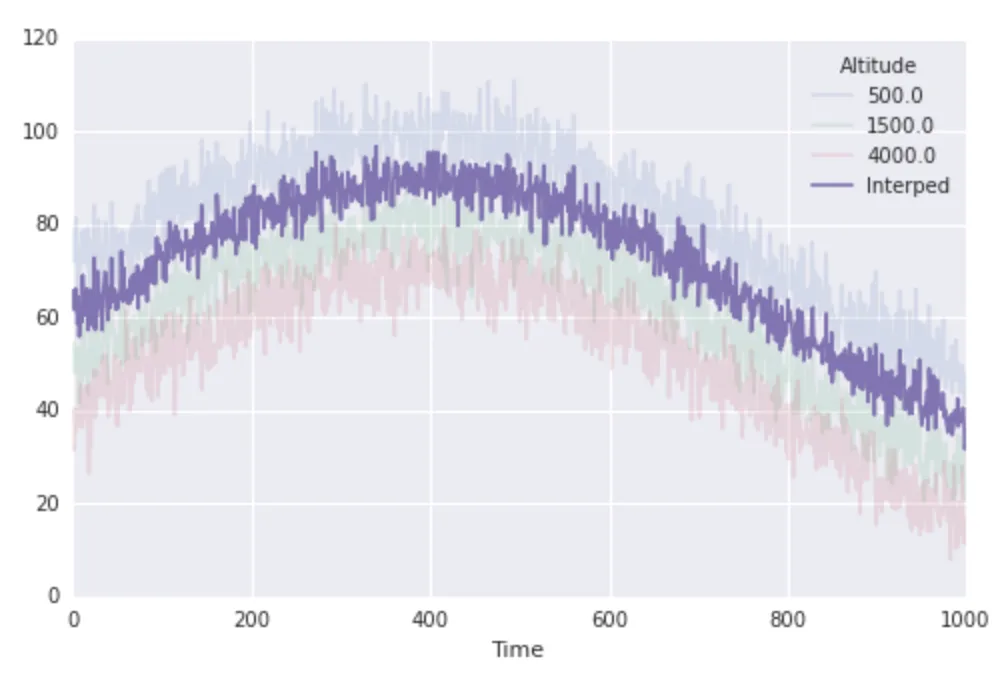
我认为这个很简单。假设我想要获取每个时间在1,000海拔处的温度。我可以使用内置的scipy插值方法:
interping_function = interp1d(altitudes, finaltemps.values)
interped_to_1000 = interping_function(1000)
fig, ax = plt.subplots(1, 1, figsize=(8, 5))
finaltemps.plot(ax=ax, alpha=0.15)
ax.plot(interped_to_1000, label='Interped')
ax.legend(loc='best', title=finaltemps.columns.name)
这很好用。现在让我们来看看速度:
%%timeit
res = interp1d(altitudes, finaltemps.values)(1000)
#-> 1000 loops, best of 3: 207 µs per loop
沿路径插值:
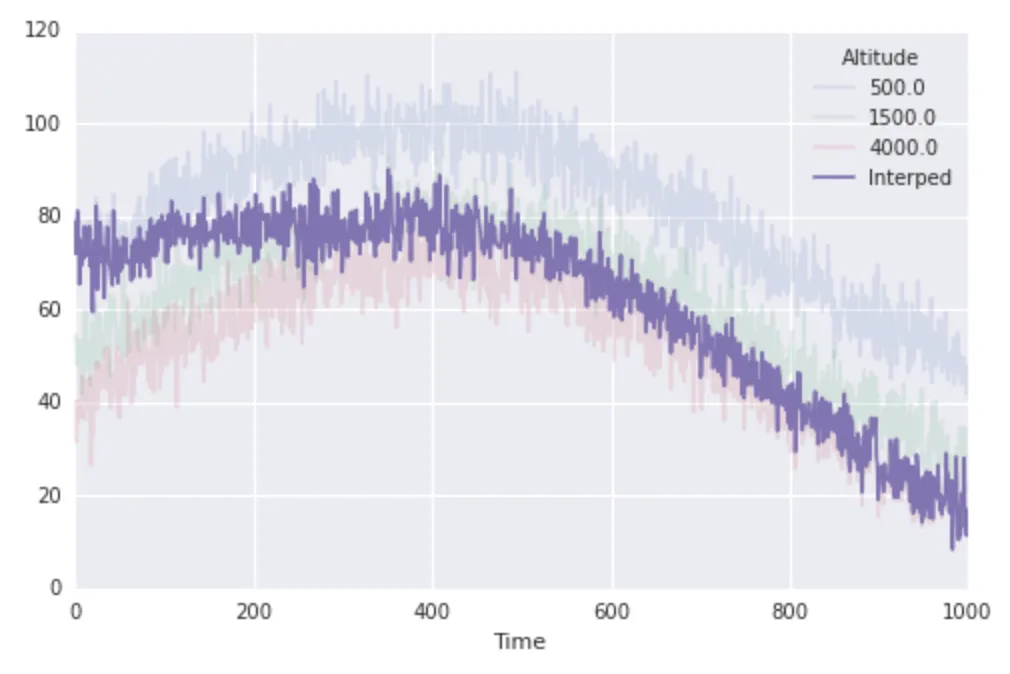
现在我有一个相关的第二个问题。假设我知道徒步旅行队的海拔随时间变化的情况,并且我想通过在时间上线性插值我的数据来计算他们(移动中)位置的温度。特别地,我知道徒步旅行队的位置的时间与我知道天气站温度的时间是相同的。我可以不费吹灰之力地做到这一点:
location = np.linspace(altitudes[0], altitudes[-1], N)
interped_along_path = np.array([interp1d(altitudes, finaltemps.values[i, :])(loc)
for i, loc in enumerate(location)])
fig, ax = plt.subplots(1, 1, figsize=(8, 5))
finaltemps.plot(ax=ax, alpha=0.15)
ax.plot(interped_along_path, label='Interped')
ax.legend(loc='best', title=finaltemps.columns.name)
这很好地解决了问题,但需要注意的是上面的关键行使用列表推导式来隐藏大量的工作。在之前的情况下,scipy为我们创建了一个单一的插值函数,并在大量数据上评估它一次。而在这种情况下,scipy实际上正在构建N个单独的插值函数,并对少量数据进行每次评估。这感觉本质上是低效的。这里潜藏着一个for循环(在列表推导式中),而且这感觉有些臃肿。
毫不奇怪,这比之前的情况要慢得多:
%%timeit
res = np.array([interp1d(altitudes, finaltemps.values[i, :])(loc)
for i, loc in enumerate(location)])
#-> 10 loops, best of 3: 145 ms per loop
因此,第二个示例的运行速度比第一个慢了1,000倍。也就是说,这符合“制作线性插值函数”步骤需要的时间在第二个示例中发生了1,000次,而在第一个示例中只发生了一次。
那么问题来了:有没有更好的方法来解决第二个问题?例如,是否有一种好的方法可以使用二维插值(也许可以处理当徒步旅行队位置已知的时间并不是温度被采样的时间的情况)?或者是否有一种特别巧妙的方法来处理时间对齐的情况?还是其他方法?