我想知道是否有一种方法可以在Python中绘制嵌套的networkx图形。按照networkx文档中描述的方式,我可以成功地使用nx.draw_(...)方法调用来绘制这些图形,但我使用它的情况需要其中一个节点本身是一个图形(想象一个房间网络,在顶层有一个房间内部区域/区域的网络在下一层)。我想使用matplotlib或类似工具来展示这个问题。如果您有任何想法,将不胜感激。
绘制嵌套的Networkx图形
4
- xyzen
2
你想要一个二维图吧? - Kirell
是的,只是一个普通的二维图,没有什么花哨的。 - xyzen
1个回答
7
编辑
您可能可以比我的原始答案更好地定义一个递归函数。以下是递归函数的大致轮廓。我的下面的答案提供了一种不太优雅的方法,可以轻松调整以适应特定情况,但如果您经常执行此操作,则可能需要使用此递归版本。
def recursive_draw(G,currentscalefactor=0.1,center_loc=(0,0),nodesize=300, shrink=0.1):
pos = nx.spring_layout(G)
scale(pos,currentscalefactor) #rescale distances to be smaller
shift(pos,center_loc) #you'll have to write your own code to shift all positions to be centered at center_loc
nx.draw(G,pos=pos, nodesize=nodesize)
for node in G.nodes_iter():
if type(node)==Graph: # or diGraph etc...
recursive_draw(node,currentscalefactor=shrink*currentscalefactor,center_loc=pos[node], nodesize = nodesize*shrink, shrink=shrink)
如果有人创建了递归函数,请将其作为单独的答案添加,并给我评论。 我会从这个答案中指向它。
原始回答 这是第一次尝试(我希望今天结束前能编辑完整个答案,但我认为这可以让你完成大部分工作):
import networkx as nx
import pylab as py
G = nx.Graph()
H = nx.Graph()
H.add_edges_from([(1,2), (2,3), (1,3)])
I = nx.Graph()
I.add_edges_from([(1,3), (3,2)])
G.add_edge(H,I)
Gpos = nx.spring_layout(G)
Hpos = nx.spring_layout(H)
Ipos = nx.spring_layout(I)
scalefactor = 0.1
for node in H.nodes():
Hpos[node] = Hpos[node]*scalefactor + Gpos[H]
for node in I.nodes():
Ipos[node] = Ipos[node]*scalefactor + Gpos[I]
nx.draw_networkx_edges(G, pos = Gpos)
nx.draw_networkx_nodes(G, pos = Gpos, node_color = 'b', node_size = 15000, alpha = 0.5)
nx.draw(H, pos = Hpos, with_labels = True)
nx.draw(I, pos = Ipos, with_labels = True)
py.savefig('tmp.png')
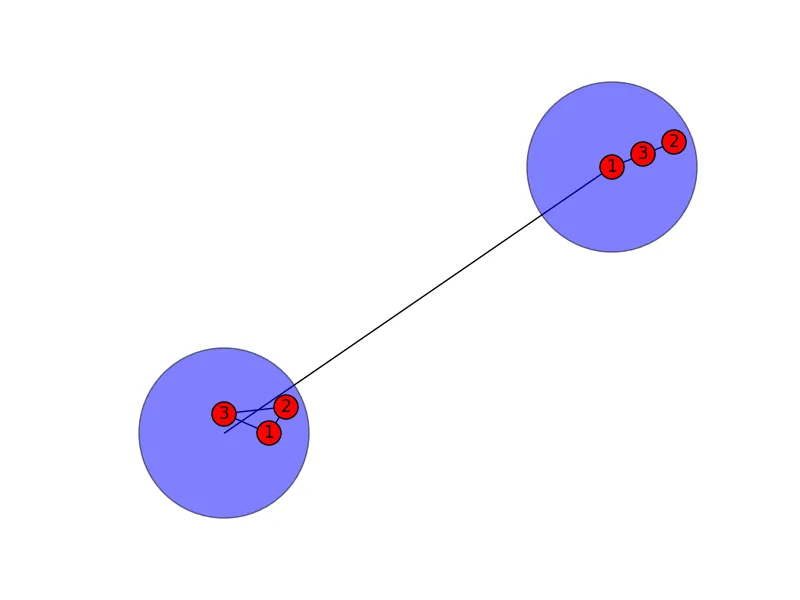
我认为你需要做的主要额外工作是将每个子节点居中。这需要为每个子图标识xmin、xmax、ymin和ymax并进行调整。您可能还想尝试调整比例因子。
- Joel
1
谢谢Joel,我没有想到那个。这已经足够让我开始了 :) 除非在接下来的一天左右出现其他/更干净的解决方案,否则我很可能会将其标记为答案! - xyzen
网页内容由stack overflow 提供, 点击上面的可以查看英文原文,
原文链接
原文链接