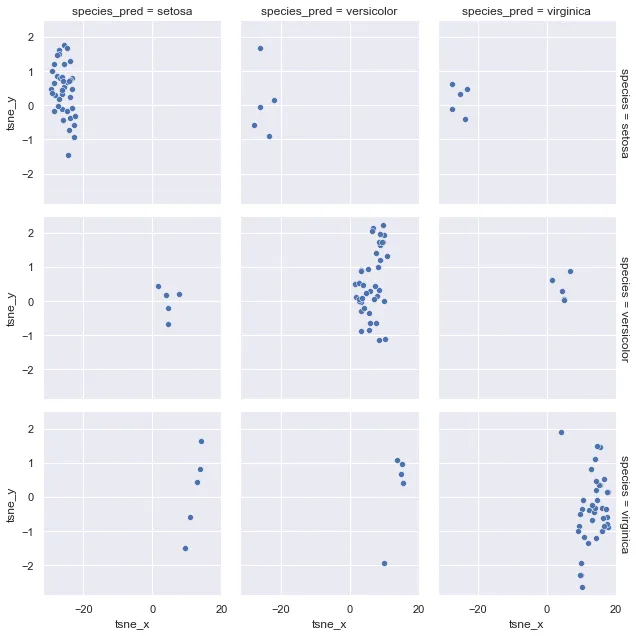
我有一个类似这样的数据框:
label predicted F1 F2 F3 .... F40
major minor 2 1 4
major major 1 0 10
minor patch 4 3 23
major patch 2 1 11
minor minor 0 4 8
patch major 7 3 30
patch minor 8 0 1
patch patch 1 7 11
我有一个label,它是id的真实标签(由于不相关,未显示),以及predicted标签和我的df中约40个特征的集合。
这个想法是将这40个特征转换为2个维度,并将它们可视化为真实值与预测值。我们有9种情况适用于所有三个标签major,minor和patch与它们的预测。
使用PCA,无法捕获太多方差,只有2个组件,我不确定如何将PCA值与原始df中的标签和预测映射为整体。实现这一点的方法是将所有情况分成9个数据框并实现结果,但这不是我要寻找的。
是否有其他方法可以减少和可视化给定的数据?任何建议都将不胜感激。