例如,对于一个包含50K行数据和窗口大小为50的数据集,对于每一行,我需要取最后50行,删除前三个和后三个值(窗口大小的5%,向上取整),并获取其余44个值的平均值。
目前,对于每一行,我都会切片以获取窗口,对窗口进行排序,然后再次切片以修剪它。这种方法可以工作,但速度较慢,必须有更有效的方法。
示例:
[10,12,8,13,7,18,19,9,15,14] # data used for example, in real its a 50k lines df
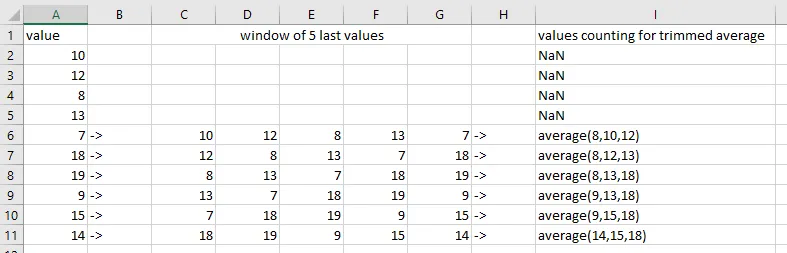 对于窗口大小为5的情况,我们查看最后5行,对它们进行排序并丢弃1个顶部行和1个底部行(5%的5 = 0.25,向上取整为1)。然后我们计算剩余中间行的平均值。
对于窗口大小为5的情况,我们查看最后5行,对它们进行排序并丢弃1个顶部行和1个底部行(5%的5 = 0.25,向上取整为1)。然后我们计算剩余中间行的平均值。
生成此示例数据框的代码
pd.DataFrame({
'value': [10, 12, 8, 13, 7, 18, 19, 9, 15, 14],
'window_of_last_5_values': [
np.NaN, np.NaN, np.NaN, np.NaN, '10,12,8,13,7', '12,8,13,7,18',
'8,13,7,18,19', '13,7,18,19,9', '7,18,19,9,15', '18,19,9,15,14'
],
'values that are counting for average': [
np.NaN, np.NaN, np.NaN, np.NaN, '10,12,8', '12,8,13', '8,13,18',
'13,18,9', '18,9,15', '18,15,14'
],
'result': [
np.NaN, np.NaN, np.NaN, np.NaN, 10.0, 11.0, 13.0, 13.333333333333334,
14.0, 15.666666666666666
]
})
window_size = 5
outliers_to_remove = 1
for index in range(window_size - 1, len(df)):
current_window = df.iloc[index - window_size + 1:index + 1]
trimmed_mean = current_window.sort_values('value')[
outliers_to_remove:window_size - outliers_to_remove]['value'].mean()
# save the result and the window content somewhere
关于DataFrame、列表和NumPy数组的说明
仅通过将数据从DataFrame转换为列表,使用相同算法即可获得3.5倍的速度提升。有趣的是,使用NumPy数组也可以获得几乎相同的速度提升。然而,必须有更好的方式来实现这一点,以实现数量级的提升。