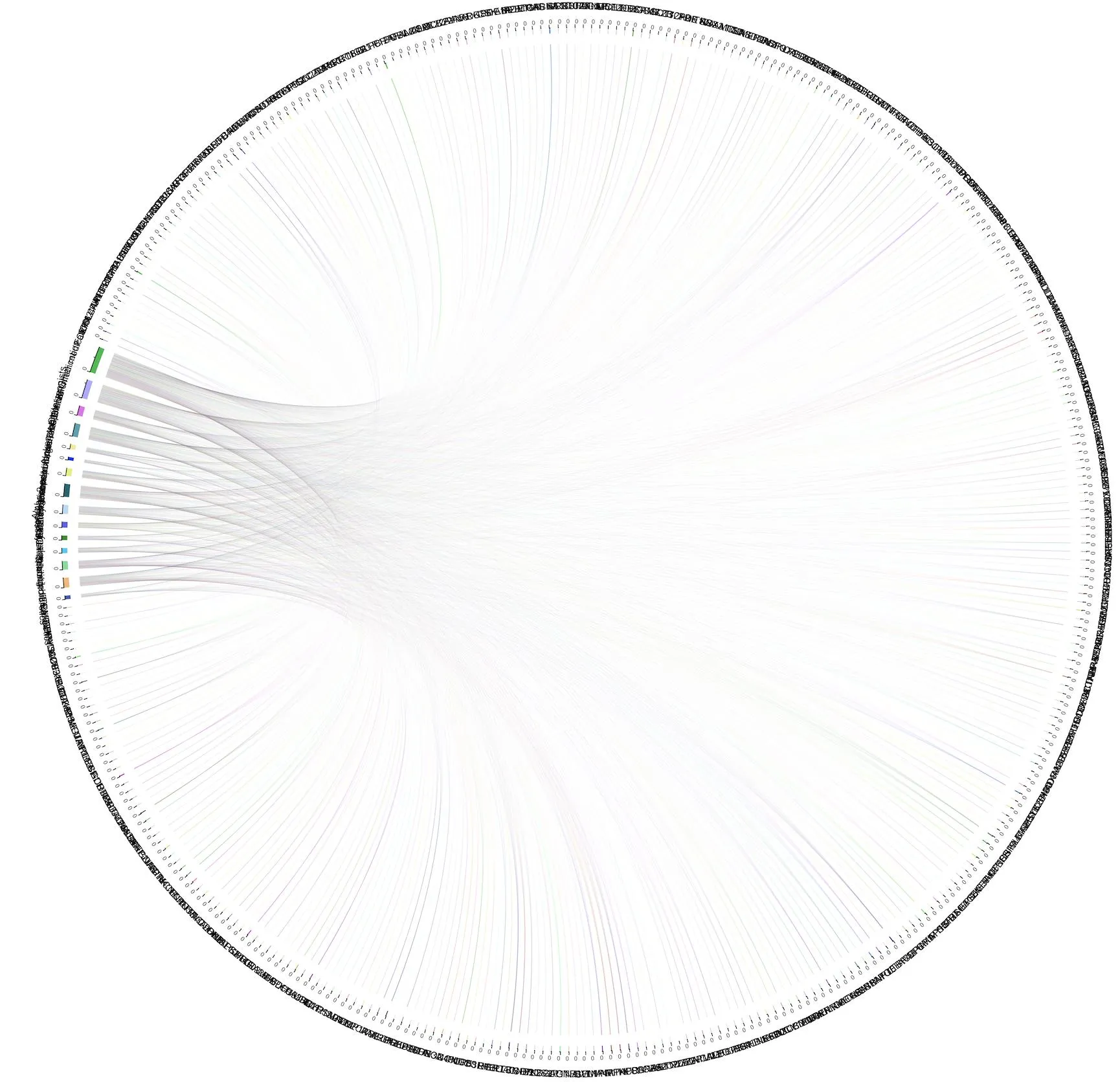
我从未制作过这样的图表,所以很抱歉这可能是一个基本问题,但我无法理解如何制作弦图,并具体使外部区域成为我的列标题(药物机制),内部连接成为行(基因),其中不需要在绘图中命名,因为太多了。
我的数据是一些基因行通过0或1标记与药物机制列互动。
例如,我的数据子集如下:
我的数据是一些基因行通过0或1标记与药物机制列互动。
例如,我的数据子集如下:
Gene Diuretic Beta_blocker ACE_inhibitor
Gene1 1 0 0
Gene2 0 0 1
Gene3 1 1 1
Gene4 0 1 1
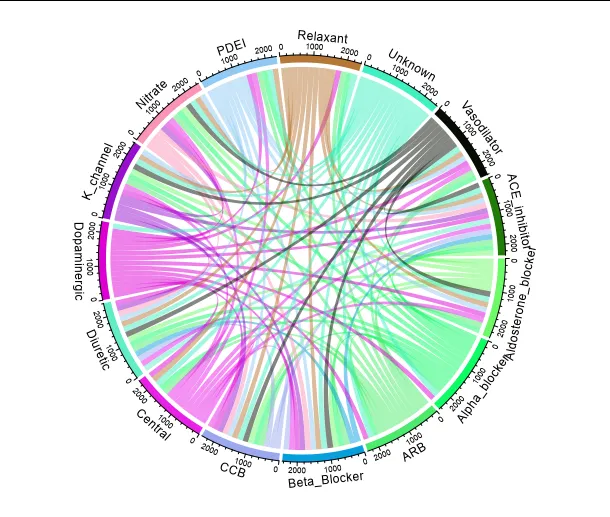
我的总数据实际上是 700 个基因,涉及 15 列药物机制,所有的值都是零和一。目前我只是在创建一个切尔德图,具体细节如下:
df <- fread('df.csv')
df[is.na(df)] <- 0
df <- df %>% data.frame %>% set_rownames(.$Gene) %>% dplyr::select(-Gene)
mt <- as.matrix(df)
circos.par(gap.degree = 0.9) #set this as I was otherwise getting an error with my total data
chordDiagram(mt, transparency = 0.5)
在尝试将此图形设置为仅有15个部分(甚至只是尝试使每个部分具有列名称)时,我遇到了各种错误。
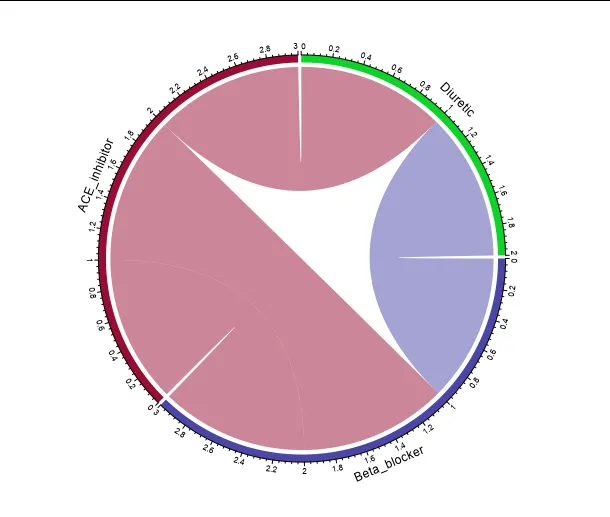
有没有一种方法可以绘制弦图,使得每个部分代表每一列?对于具有交互作用(数据中的1)的基因/行,以及任何其他部分,都可以在弦图中显示?我不需要看到基因名称,我只想可视化我的列/部分之间的重叠程度。
示例输入数据(我的问题是尝试使其每个列仅有3个部分,以显示它们的重叠):
df <- structure(list(Gene = c("Gene1", "Gene2", "Gene3", "Gene4"),
Diuretic = c(1L, 0L, 1L, 0L), Beta_blocker = c(0L, 0L, 1L,
1L), ACE_inhibitor = c(0L, 1L, 1L, 1L)), row.names = c(NA,
-4L), class = c("data.table", "data.frame")