如果你想在保留其他现有社区的同时删除某些社区,你不能创建一个由你想要保留的顶点组成的诱导子图并在子图上进行聚类,因为结果社区很可能会改变。
一个可行的方法是手动对社区对象进行子集化。
另外,如果你想绘制原始图形和社区以及新的社区,并且在所有地方都保持相同的颜色,则需要执行几个额外的步骤。
Original Answer: "最初的回答"
suppressPackageStartupMessages(library(igraph))
set.seed(123)
g <- erdos.renyi.game(50, 0.05)
c <- cluster_louvain(g)
l <- layout_with_fr(g)
c_keep_ids <- as.numeric(names(sizes(c)[sizes(c) >= 5]))
c_keep_v_idxs <- which(c$membership %in% c_keep_ids)
g_sub <- induced_subgraph(g, V(g)[c_keep_v_idxs])
c_sub <- c
c_sub$names <- c$names[c_keep_v_idxs]
c_sub$membership <- c$membership[c_keep_v_idxs]
c_sub$vcount <- length(c_sub$names)
c_sub$modularity <- modularity(g_sub, c_sub$membership, E(g_sub)$weight)
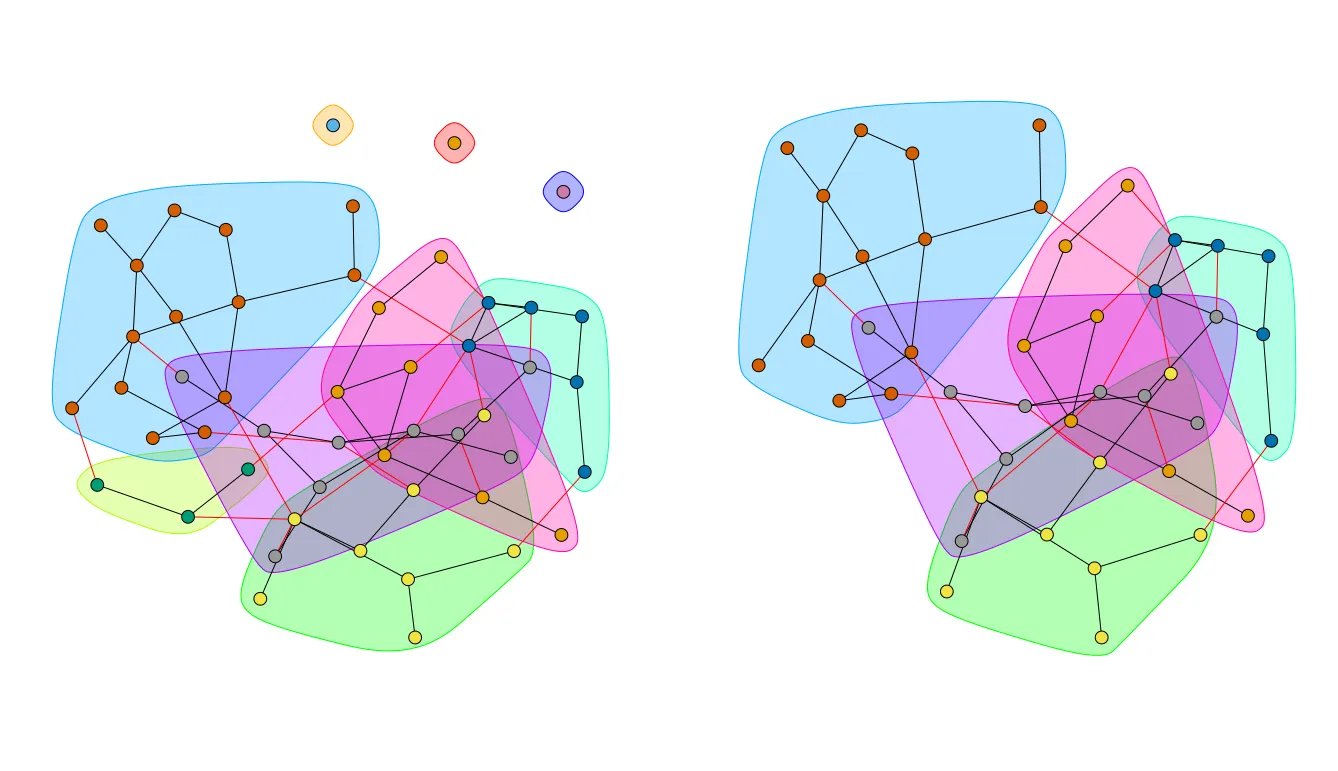
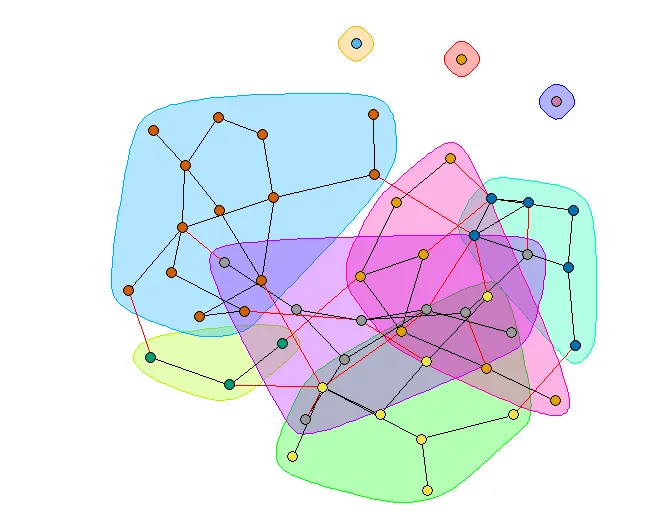
par(mfrow = c(1, 2))
plot(c, g,
layout = l,
vertex.label = NA,
vertex.size = 5
)
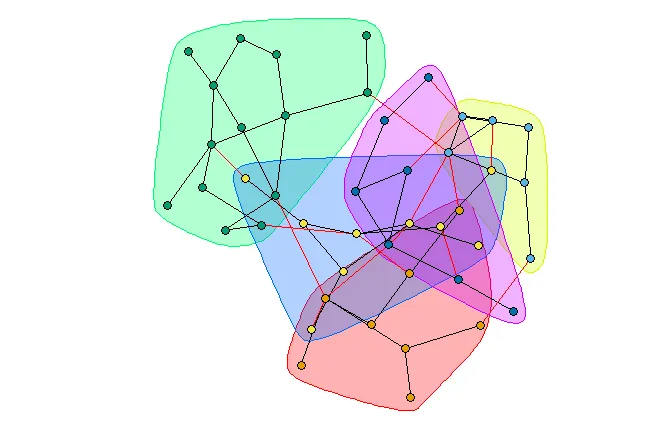
plot(c_sub, g_sub,
col = membership(c)[c_keep_v_idxs],
layout = l[c_keep_v_idxs, ],
mark.border = rainbow(length(communities(c)), alpha = 1)[c_keep_ids],
mark.col = rainbow(length(communities(c)), alpha = 0.3)[c_keep_ids],
vertex.label = NA,
vertex.size = 5
)
par(mfrow = c(1, 1))

lou的样本? - gos