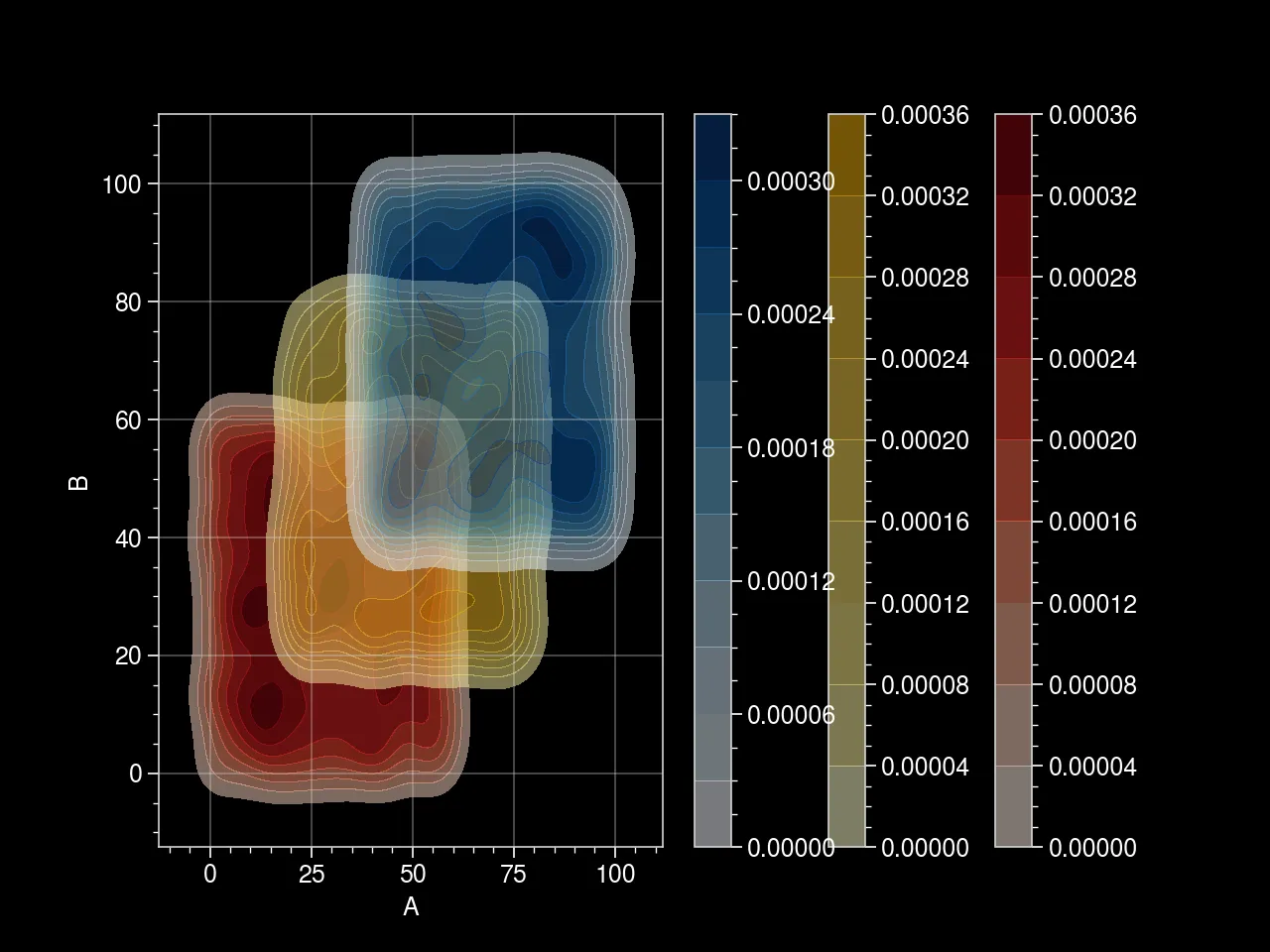
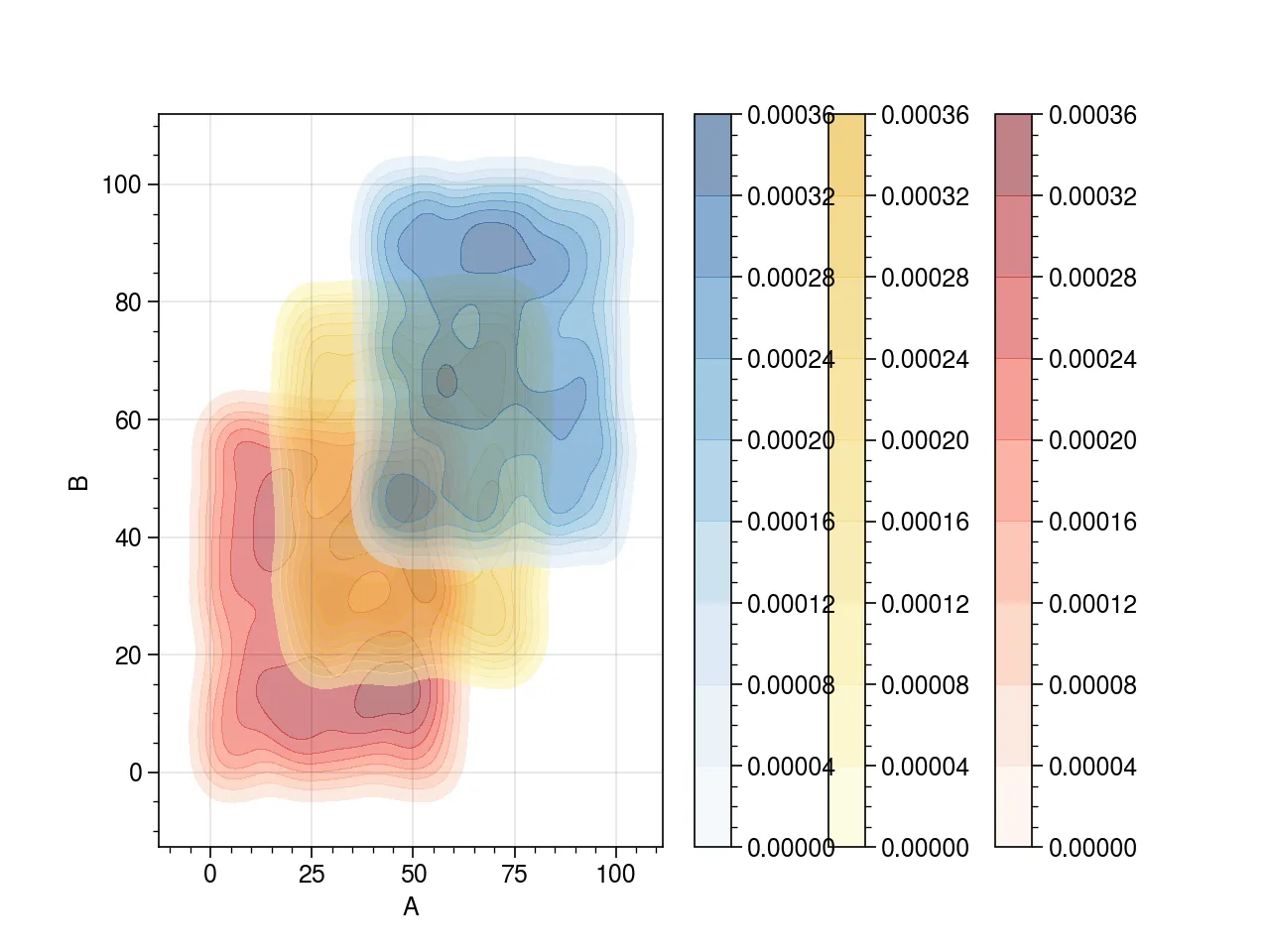
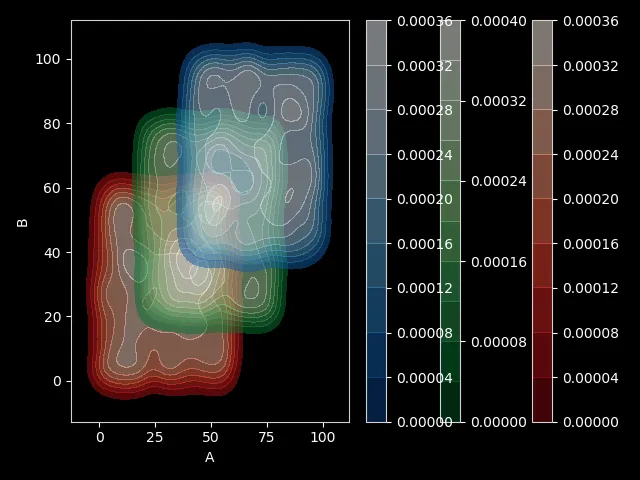
我有三个数据集(df1,df2,df3,这里是随机生成的示例),我想在黑色背景上一起绘制联合核密度图。我不喜欢联合kde的重叠部分,因为当白色部分(最低密度)与黑色背景重叠时,它真的很突出。相比之下,这在白色背景下看起来很好(将在底部包括以进行比较),但我需要它成为黑色背景。
改进方法的一个想法可能是:
- 颠倒颜色条,使最低密度为暗色,最高密度为亮色。
有人知道如何做到这一点或如何使其更好吗?
我不知道从哪里开始查找,但我在seaborn的GitHub上发现了这个closed issue,其中有关于阴影的讨论。
import numpy as np
import seaborn as sns
import pandas as pd
import matplotlib.pyplot as plt
# Uncomment for black background figure
plt.rcParams.update({
"grid.color": "white",
'hatch.color': 'k',
"lines.color": "white",
"patch.edgecolor": "white",
'patch.facecolor': ([0, 1, 1]),
'grid.alpha': 0.4,
"text.color": "lightgray",
"axes.facecolor": "black",
"axes.edgecolor": "lightgray",
"axes.labelcolor": "white",
"xtick.color": "white",
"ytick.color": "white",
"grid.color": "lightgray",
"figure.facecolor": "black",
"figure.edgecolor": "black",
"savefig.facecolor": "black",
"savefig.edgecolor": "black"})
df1 = pd.DataFrame(np.random.randint(0,60,size=(1500, 4)), columns=list('ABCD'))
df2 = pd.DataFrame(np.random.randint(20,80,size=(1500, 4)), columns=list('ABCD'))
df3 = pd.DataFrame(np.random.randint(40,100,size=(1500, 4)), columns=list('ABCD'))
f, axs = plt.subplots()
# Draw density plots
axs = sns.kdeplot(df1.A, df1.B, alpha=0.5,
cmap="Reds", shade=True, shade_lowest=False, cbar=True)
axs = sns.kdeplot(df2.A, df2.B, alpha=0.5,
cmap="Oranges", shade=True, shade_lowest=False, cbar=True)
axs = sns.kdeplot(df3.A, df3.B, alpha=0.5,
cmap="Blues", shade=True, shade_lowest=False, cbar=True)
“Reds_r”不是名称的有效值;支持的值包括“538”、“accent”、“acton”、“algae”、“amp”、“balance”、“bamako”…我将继续迭代以找出原因。不过你的图看起来很好,正是我想要的。 - JAG20243.1.3,Python 版本为3.7.6。 - Salvatorereds(全小写)。你可以尝试使用reds_r。 - Salvatorepip install matplotlib==3.1.3切换到您的matplotlib版本,就可以使用颜色映射:reds_r,blues_r等!非常感谢。是的,我同意@TrentonMcKinney的充足间距,它与plt.figure(figsize=(10, 6), dpi=80, facecolor='w', edgecolor='k')很好地配合使用。 - JAG2024