从
余弦文档中我们可以得到以下信息:
scipy.spatial.distance.cosine(u, v):计算1-D数组之间的余弦距离。
u和
v之间的余弦距离定义为
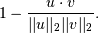
其中
u⋅v是向量
u和
v的点积。
使用上述公式,我们可以使用
`NumPy的广播能力来实现一个矢量化的解决方案,如下所示 -
dots = np.dot(A,B.T)
l2norms = np.sqrt(((A**2).sum(1)[:,None])*((B**2).sum(1)))
cosine_dists = 1 - (dots/l2norms)
minval = np.nanmin(cosine_dists,axis=1)
cosine_dists[np.isnan(cosine_dists).all(1),0] = 0
res = np.nanargmin(cosine_dists,axis=1)
运行时测试 -
In [81]: def org_app(A,B):
...: l, res, minval = A.shape[0], [], []
...: for i in xrange(l):
...: minimum = min((cosine(A[i], B[j]), j) for j in xrange(l))
...: res.append(minimum[1])
...: minval.append(minimum[0])
...: return res, minval
...:
...: def vectorized(A,B):
...: dots = np.dot(A,B.T)
...: l2norms = np.sqrt(((A**2).sum(1)[:,None])*((B**2).sum(1)))
...: cosine_dists = 1 - (dots/l2norms)
...: minval = np.nanmin(cosine_dists,axis=1)
...: cosine_dists[np.isnan(cosine_dists).all(1),0] = 0
...: res = np.nanargmin(cosine_dists,axis=1)
...: return res, minval
...:
In [82]: A = np.random.rand(400,500)
...: B = np.random.rand(400,500)
...:
In [83]: %timeit org_app(A,B)
1 loops, best of 3: 10.8 s per loop
In [84]: %timeit vectorized(A,B)
10 loops, best of 3: 145 ms per loop
验证结果 -
In [86]: x1, y1 = org_app(A, B)
...: x2, y2 = vectorized(A, B)
...:
In [87]: np.allclose(np.asarray(x1),x2)
Out[87]: True
In [88]: np.allclose(np.asarray(y1)[~np.isnan(np.asarray(y1))],y2[~np.isnan(y2)])
Out[88]: True
A = np.load('A.npy'),B = np.load('B.npy'),x1, y1 = org_app(A, B),x2, y2 = vectorized(A, B)并检查 x1 和 list(x2)。我得到了x1=[459, 571, 526, 477, 309, 498, 504, 4, 529, ..和x2=[29, 29, 29, 29, 29, 29, 29, 29, 29, 29, ...。A.npy,B.npy。 - Salvador Daliscipy.spatial.distance.cdist不会给你与余弦距离相同的结果。要验证,请查看这两种方法的最小距离,而不仅仅是最小参数。 - Divakarscipy.spatial.distance.cdist,对于随机输入返回相同的结果,但速度较慢。 我无法访问A.npy或B.npy,因此无法在那里检查结果。 如果您使用我发布的内容,则可能需要自己调整它以使其按照您想要的方式处理NaN。 - user2034412