看一下以下情况:
好的,根据这个,我在R中拟合了上面列出的模型(不过,我不确定这些模型是否正确)。
好的,现在我的兴趣是进行涉及以下比较的方差分量测试:
模型2与模型1的比较 模型3与模型2的比较 模型4与模型2的比较
请参见下面的代码:
然而,有些问题出现了(我怀疑是在我的模型4的规范中主要存在问题)。 事情是这样的:根据我想要得出的结果,涉及模型2和模型4之间比较的p值应该为
请参考: 因此,在使用
以下是随机效应协方差结构的结果:
在这个主题中:具有负方差的混合效应模型中提到了
查看随机效应协方差结构值:
然而,问题仍然存在。因此,根据帖子中的初始信息,我有一种感觉我的模型在R中没有正确实现,因为方差成分的测试结果与预期不一致。是否有任何解决方法?
举个例子,我将使用同样的情况,但考虑到R中的Orthodont数据。
library(nlme)
model1 <- lm(Y ~ Treatm * VarT, data = datarats)
model2 <- lme(Y ~ Treatm * VarT, data = datarats, random = ~ 1|RAT, method = "ML")
model3 <- lme(Y ~ Treatm * VarT, data = datarats, random = list(RAT = pdDiag(~ VarT)), method = "ML")
model4 <- lme(Y ~ Treatm * VarT, data = datarats, random = ~ 1 + VarT | RAT, method = "ML")
好的,现在我的兴趣是进行涉及以下比较的方差分量测试:
模型2与模型1的比较 模型3与模型2的比较 模型4与模型2的比较
请参见下面的代码:
library(varTestnlme)
library(EnvStats)
library(lmeInfo)
Comp1 <- varCompTest(model2, model1, pval.comp = "both")
summary(Comp1)
print.htestEnvStats(Comp1)
------
Comp2 <- varCompTest(model3, model2, pval.comp = "both")
summary(Comp2)
print.htestEnvStats(Comp2)
------
Comp3 <- varCompTest(model4, model2, pval.comp = "both")
summary(Comp3)
print.htestEnvStats(Comp3)
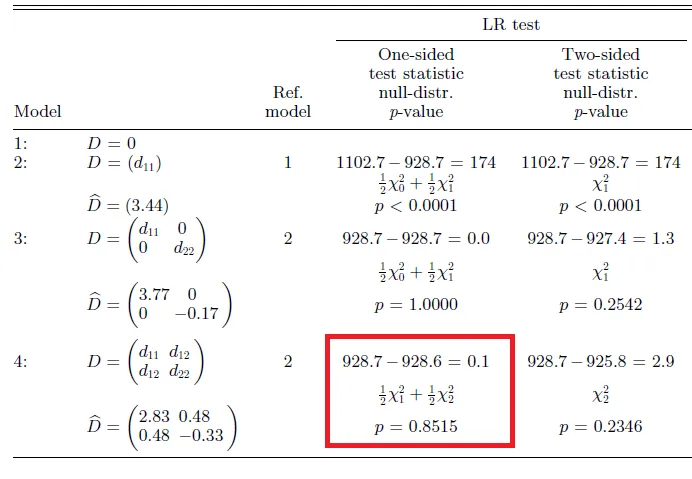
然而,有些问题出现了(我怀疑是在我的模型4的规范中主要存在问题)。 事情是这样的:根据我想要得出的结果,涉及模型2和模型4之间比较的p值应该为
p-value = 0.85
((1 - pchisq(0.1,1))/2 + (1 - pchisq(0.1,2))/2,其中0.1是两个模型的loglik之间的差异。请参考: 因此,在使用
varTestnlme包进行测试时,我得到了p-value = 1,即模型2和模型4是相等的(两个模型的logLik()之间的差异为零)。
请查看结果:summary(Comp3)
Variance components testing in mixed effects models
Testing that:
variance of the random effect associated to VarT is equal to 0
against the alternative that:
variance of the random effect associated to VarT > 0
Likelihood ratio test statistic:
LRT = -1.759843e-07
Limiting distribution:
mixture of 2 chi-bar-square distributions with degrees of freedom 1 2
associated (exact) weights: 0.5 0.5
p-value of the test:
from exact weights: 1
以下是随机效应协方差结构的结果:
> VarCorr(model2)
RAT = pdLogChol(1)
Variance StdDev
(Intercept) 3.289539 1.813709
Residual 1.423777 1.193221
> VarCorr(model4)
RAT = pdLogChol(1 + VarT)
Variance StdDev Corr
(Intercept) 3.289539e+00 1.813708744 (Intr)
VarT 1.492819e-08 0.000122181 0
Residual 1.423777e+00 1.193221261
在这个主题中:具有负方差的混合效应模型中提到了
nlme包在模型中存在负方差的限制,并且在上面的打印中可以看到D矩阵的斜率变化有一个负数。然而,在同一篇文章中:具有负方差的混合效应模型中提到解决这个问题的方法之一是考虑复合对称方差结构,这将允许组内出现负相关。因此,我对模型进行了如下调整:model2 <- lme(Y ~ Treatm * VarT, data = datarats, random = ~ 1|RAT,
correlation = corSymm(form = ~1),
method = "ML")
model3 <- lme(Y ~ Treatm * VarT, data = datarats, random = list(RAT = pdDiag(~ VarT)),
correlation = corSymm(form = ~1),
control = lmeControl(opt = "optim", optCtrl = list(method = "BFGS")),
method = "ML")
model4 <- lme(Y ~ Treatm * VarT, data = datarats, random = ~ 1 + VarT | RAT,
correlation = corSymm(form = ~1),
control = lmeControl(opt = "optim", optCtrl = list(method = "BFGS")),
method = "ML")
查看随机效应协方差结构值:
> VarCorr(model2)
RAT = pdLogChol(1)
Variance StdDev
(Intercept) 3.338813 1.827242
Residual 1.417336 1.190519
> VarCorr(model3)
RAT = pdDiag(VarT)
Variance StdDev
(Intercept) 3.370298164 1.83583718
VarT 0.001353776 0.03679369
Residual 1.384231329 1.17653361
> VarCorr(model4)
RAT = pdLogChol(1 + VarT)
Variance StdDev Corr
(Intercept) 3.372970029 1.83656474 (Intr)
VarT 0.002481283 0.04981247 0.019
Residual 1.375129804 1.17265929
然而,问题仍然存在。因此,根据帖子中的初始信息,我有一种感觉我的模型在R中没有正确实现,因为方差成分的测试结果与预期不一致。是否有任何解决方法?
举个例子,我将使用同样的情况,但考虑到R中的Orthodont数据。
data(Orthodont)
library(nlme)
library(varTestnlme)
library(EnvStats)
library(lmeInfo)
model11 <- lm(distance ~ Sex * age, data = Orthodont)
model22 <- lme(distance ~ Sex * age, data = Orthodont, random = ~ 1|Subject,
method = "ML")
model33 <- lme(distance ~ Sex * age, data = Orthodont, random = list(Subject = pdDiag(~ age)),
method = "ML")
model44 <- lme(distance ~ Sex * age, data = Orthodont, random = ~ 1 + age | Subject,
method = "ML")
Comp11 <- varCompTest(model22, model11, pval.comp = "both")
summary(Comp11)
print.htestEnvStats(Comp11)
Comp22 <- varCompTest(model33, model22, pval.comp = "both")
summary(Comp22)
print.htestEnvStats(Comp22)
Comp33 <- varCompTest(model44, model22, pval.comp = "both")
summary(Comp33)
print.htestEnvStats(Comp33)

datarats? - swihartdput(datarats)的输出吗?我的笔记本电脑上被阻止访问Google Drive。 - Stéphane Laurentdput(datarats)的输出吗?Google Drive 在我的笔记本上被封锁了。 - Stéphane Laurent