我的变量是在随机分块和子采样设计的测量下进行的,其中我的处理为23个接种物。我有3个完整的块和每块6个样本。示例数据框包括4个响应变量(LH、REN、FTT、DFR)、Accesion(处理)、Bloque(块编号)和Plot(用于解释子采样的变量)。数据的头部如下:
Plot Accesion Bloque LH REN FTT DFR
1 221 22 1 20.6 1127 23 88
2 221 22 1 20.5 1638 20 88
3 221 22 1 24.5 1319 16 88
4 221 22 1 21.4 960 17 88
5 221 22 1 25.7 1469 18 88
6 221 22 1 25.8 1658 21 88
因此,在各种转换(对数、boxcox、幂等等)后,几乎所有的100个响应变量的数据都是非正态和异方差的。大多数变量显示出具有不同方差的卡方或泊松分布。
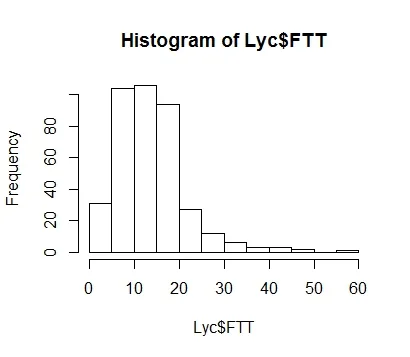
到目前为止,我一直在运行一个广义线性模型-使用Poisson作为响应变量的效应,我正在使用以下代码:glmer()。
FTTglme = glmer(FTT ~ Accesion + Bloque + (1|Plot), data = Lyc,
family=poisson(link="identity"))
残差不服从shapiro.test()的正态分布。我认为这是因为残差中存在异方差性。通过根据Accesion绘制残差箱线图可以看出方差之间的差异:
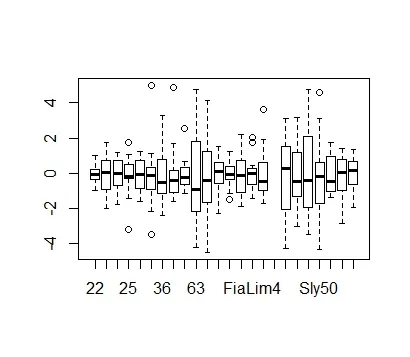 预计在植物群体之间会存在异方差性,但我知道它可以在glme内进行建模。我已经调查了需要添加的代码:
预计在植物群体之间会存在异方差性,但我知道它可以在glme内进行建模。我已经调查了需要添加的代码:vf <- varIdent(form=~Accesion)
FTTglme = glmer(FTT ~ Accesion + Bloque + (1|Plot), data = Lyc,
family=poisson(link="identity"), weights = vf)
我希望不同的方差可以考虑每个接入类别。但是我一直收到错误提示:
Error in model.frame.default(data = Lyc, weights = varIdent(form = ~Accesion), :
variable lengths differ (found for '(weights)')
有人知道如何在glmer()内考虑访问之间方差差异吗?
欢迎提出任何其他分析数据的建议。
nlme和lme4语法混合使用于权重:这是行不通的... - Ben Bolker