我正在尝试对齐数组(图像)。由于非线性畸变,这些数组不共享同质坐标,因此仿射变换是不够的。
幸运的是,在数组之间找到匹配特征的坐标是微不足道的,这些坐标被用来计算初始的仿射变换。我使用坐标对之间的残差差异来拟合平滑双变量样条,然后模拟依赖于位置的偏移在x和y上的变化,以便将一个图像转换为另一个图像。
问题出现在使用geometric_transform()时的这些Splines中-虽然得到的对齐效果非常好,但速度极慢(数组大小约为50M)。
我创建了一个表示所需的x和y坐标偏移的样条(这里img1_coo是第一张图像中的x和y坐标的Nx2数组,img2_coo是经过仿射变换后的第二张图像的相同数组)。
几何变换(geometric_transform())的可调用函数如下所示:
所以我猜测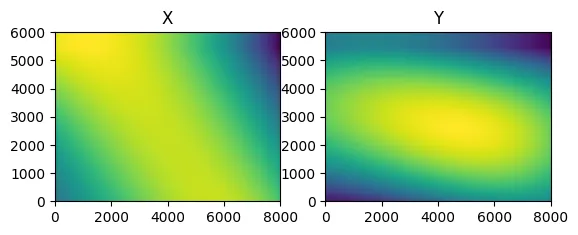 我尝试过调整插值的顺序等方法,但没有找到任何加速的方法。欢迎任何有关加速
我尝试过调整插值的顺序等方法,但没有找到任何加速的方法。欢迎任何有关加速
(我已经尝试过使用
幸运的是,在数组之间找到匹配特征的坐标是微不足道的,这些坐标被用来计算初始的仿射变换。我使用坐标对之间的残差差异来拟合平滑双变量样条,然后模拟依赖于位置的偏移在x和y上的变化,以便将一个图像转换为另一个图像。
问题出现在使用geometric_transform()时的这些Splines中-虽然得到的对齐效果非常好,但速度极慢(数组大小约为50M)。
我创建了一个表示所需的x和y坐标偏移的样条(这里img1_coo是第一张图像中的x和y坐标的Nx2数组,img2_coo是经过仿射变换后的第二张图像的相同数组)。
from scipy import interpolate
sbs_x = interpolate.SmoothBivariateSpline(img1_coo[:,0], img1_coo[:,1], img1_coo[:,0]-img2_coo[:,0])
sbs_y = interpolate.SmoothBivariateSpline(img1_coo[:,0], img1_coo[:,1], img1_coo[:,1]-img2_coo[:,1])
几何变换(geometric_transform())的可调用函数如下所示:
def apply_spline(xy):
return xy[0] - sbs_x.ev(xy[0], xy[1]), xy[1] - sbs_y.ev(xy[0], xy[1])
使用以下方法进行转换:
from scipy import ndimage
img2_data_splined = ndimage.geometric_transform(img2_data, apply_spline)
这在一个大小为50M的数组上需要约10分钟时间。我发现使用50M大小的数组评估SmoothBivariateSpline.ev(x,y)非常快:
xx, yy = np.meshgrid(np.arange(8000), np.arange(6000))
%timeit sbs_x.ev(xx,yy)
6.78 s ± 43.9 ms per loop (mean ± std. dev. of 7 runs, 1 loop each)
所以我猜测
geometric_transform()在单独调用每个坐标时速度较慢。为了可视化,这是我正在纠正的样条地图(颜色从〜-1像素到+5像素移位)。
 我尝试过调整插值的顺序等方法,但没有找到任何加速的方法。欢迎任何有关加速
我尝试过调整插值的顺序等方法,但没有找到任何加速的方法。欢迎任何有关加速geometric_transform()或执行图像配准和处理复杂几何/扭曲的其他实现的帮助。(我已经尝试过使用
PolynomialTransform的skimage.warp,但对齐不太好,而且速度也相当慢,但不像geometric_transform()那么慢)
apply_spline函数中使用map_coordinates,但是调用该函数5000万次的代价仍然存在。您的第一个答案解决了我的问题(现在只需要大约10-15秒!)。我喜欢第二个答案的想法,也许以后会考虑。 - Jdog