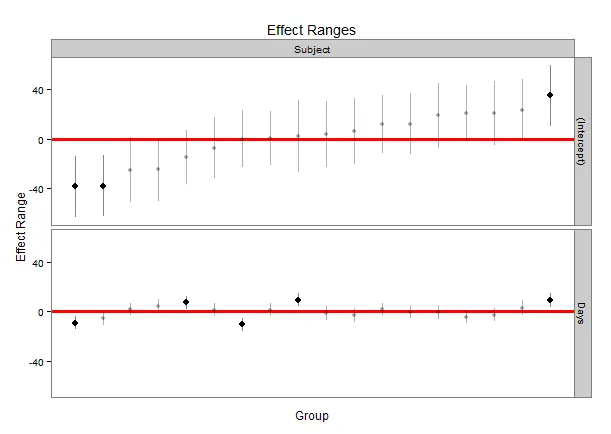
qqmath函数使用lmer软件包的输出来生成随机效应的优秀毛虫图。也就是说,qqmath非常适合绘制分层模型中拦截的估计值及其误差的图表。下面是使用lme4软件包中名为Dyestuff的内置数据的lmer和qqmath函数示例。该代码将生成分层模型以及使用ggmath函数生成漂亮的图表。
library("lme4")
data(package = "lme4")
# Dyestuff
# a balanced one-way classiï¬cation of Yield
# from samples produced from six Batches
summary(Dyestuff)
# Batch is an example of a random effect
# Fit 1-way random effects linear model
fit1 <- lmer(Yield ~ 1 + (1|Batch), Dyestuff)
summary(fit1)
coef(fit1) #intercept for each level in Batch
# qqplot of the random effects with their variances
qqmath(ranef(fit1, postVar = TRUE), strip = FALSE)$Batch
代码的最后一行生成了一个非常好看的图,显示了每个截距与估计误差的情况。但是格式化qqmath函数似乎非常困难,我一直在努力格式化这个图表。我想出了一些问题,无法回答,而且我认为如果其他人正在使用lmer/qqmath组合,则可以从这些问题中受益:
- 是否有一种方法可以采取上面的qqmath函数并添加一些选项,例如使某些点为空心而不是填充的,或者为不同的点使用不同的颜色?例如,您可以使批次变量的A、B和C点填充,但然后使其余点为空?
- 是否可能为每个点添加轴标签(例如,沿着顶部或右侧y轴)?
- 我的数据更接近于45个截距,因此可能需要在标签之间添加间距,以便它们不互相重叠?主要是,我对区分/标记图中的点感兴趣,这似乎在ggmath函数中很麻烦/不可能。
到目前为止,在qqmath函数中添加任何其他选项都会产生错误,如果是标准图则不会出现这些错误,因此我感到迷茫。
此外,如果您觉得有更好的绘制lmer输出的截距的软件包/函数,我很乐意听取! (例如,是否可以使用dotplot完成1-3点?)
编辑: 如果可以合理地格式化,我也可以接受另一种dotplot。 我只是喜欢ggmath图表的外观,因此从一个问题开始。
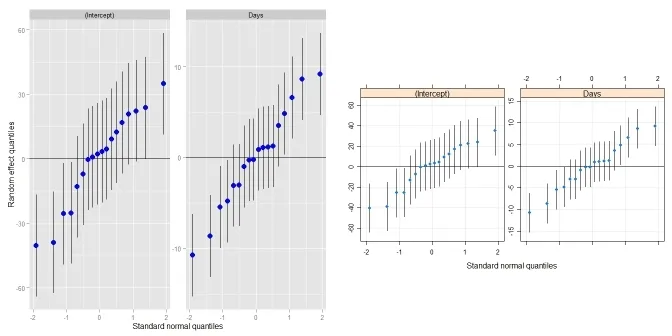
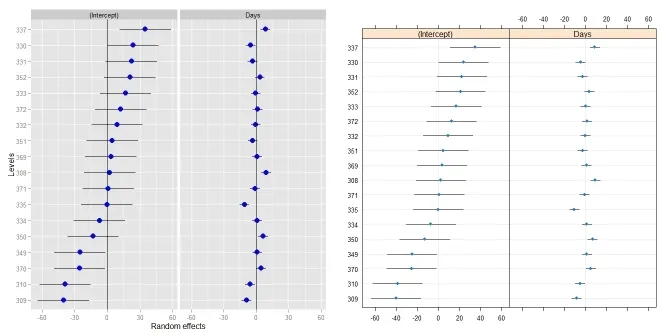
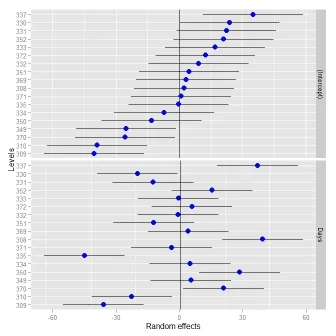
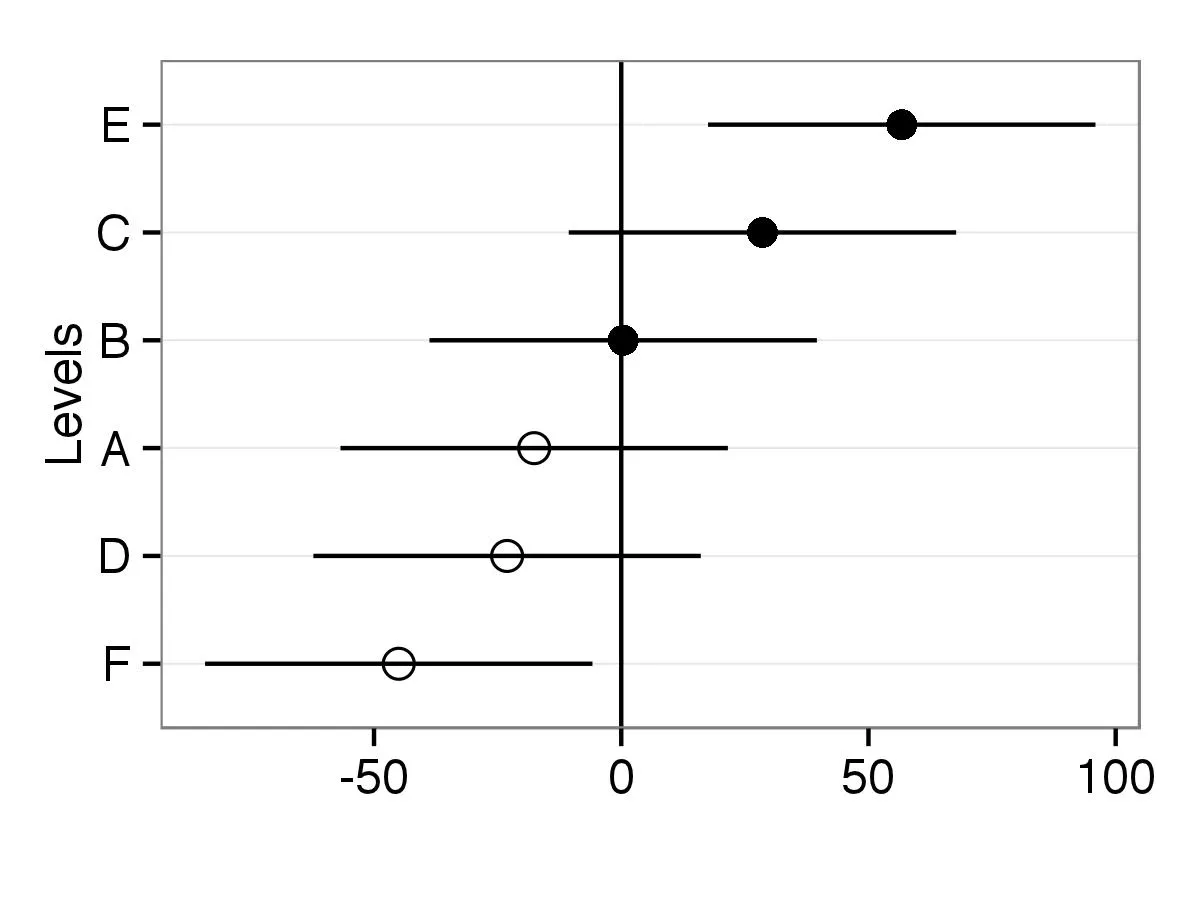
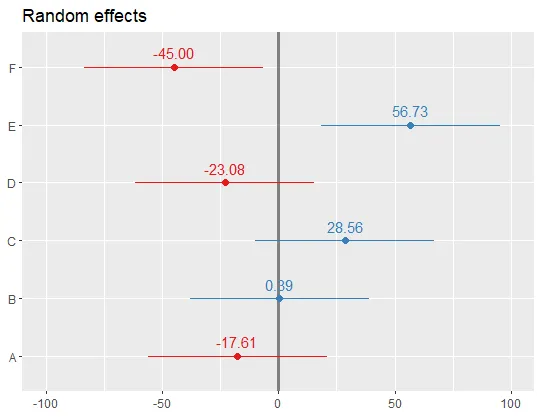
ggCaterpillar(ranef(fit, condVar=TRUE), QQ=FALSE, likeDotplot=FALSE)的调用即可。 - jay.sf