我可以使用nlme包中的gls()函数来建立没有随机效应的mod1模型。 然后,我可以使用lme()函数来建立包含随机效应的mod2模型,并通过AIC对比mod1和mod2。
mod1 = gls(response ~ fixed1 + fixed2, method="REML", data)
mod2 = lme(response ~ fixed1 + fixed2, random = ~1 | random1, method="REML",data)
AIC(mod1,mod2)
是否有类似于lme4包中gls()的东西,它允许我构建没有随机效应的mod3,并将其与使用包括随机效应的lmer()构建的mod4进行比较?
mod3 = ???(response ~ fixed1 + fixed2, REML=T, data)
mod4 = lmer(response ~ fixed1 + fixed2 + (1|random1), REML=T, data)
AIC(mod3,mod4)
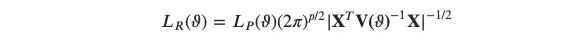
r-sig-mixed-models@r-project.org[首先订阅该列表; 你可以通过搜索找到信息/订阅页面],在任何情况下都需要提供可重现的示例(以及packageVersion("lme4")的结果)。 - Ben Bolkerr-sig-mixed-models@r-project.org上发布一个可重现的示例? - Ben Bolker