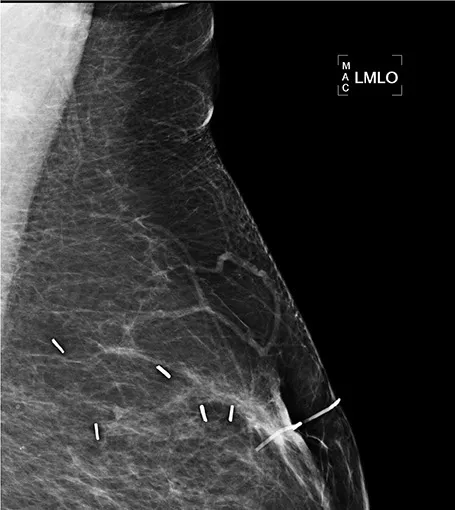
看一下您提供的图像的实际像素值,您会发现标记几乎是(99.99%)纯白色,而且在图像中没有其他地方出现,因此您可以使用简单的99.99%阈值来隔离它。
我更喜欢在命令行中使用ImageMagick,所以我会这样做:
convert sample.dcm -threshold 99.99% -negate mask.png

convert sample.dcm mask.png -compose darken -composite result.jpg
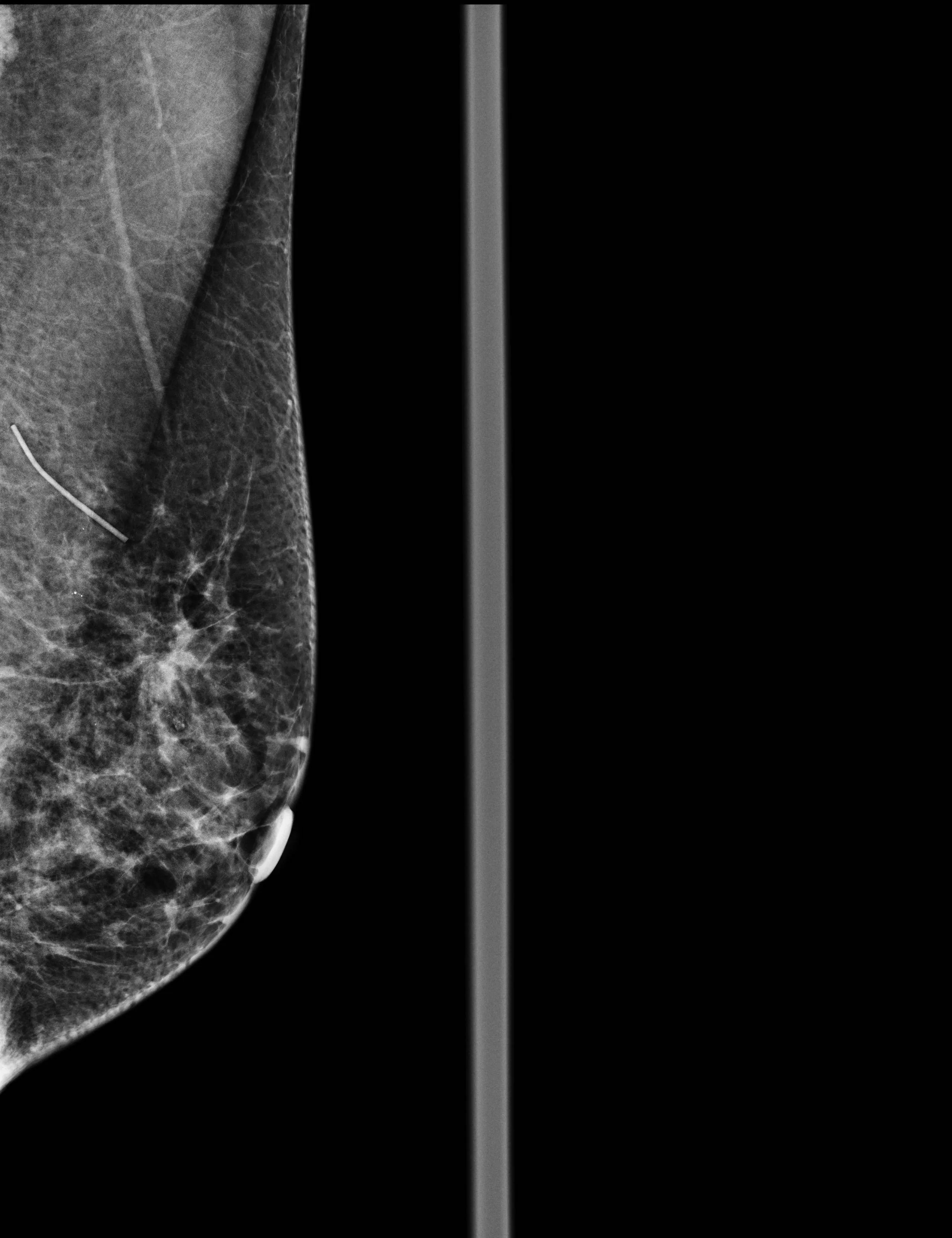
当然,如果样本图像不具有代表性,您可能需要更加努力。让我们看看这个...
如果简单阈值对您的图像无效,我会尝试使用“命中与失误形态学”技术。基本上,您将图像阈值设置为纯黑和白 - 大约在90%左右,然后寻找特定的形状,例如标签上的角标记。因此,如果我们想要查找黑色背景上白色矩形的左上角,并且我们使用0表示“此像素必须是黑色”,1表示“此像素必须是白色”,-表示“我们不关心”,我们将使用此模式:
0 0 0 0 0
0 1 1 1 1
0 1 - - -
0 1 - - -
0 1 - - -
希望您能在左上角看到一个白色矩形。在终端中,它应该是这样的:
convert sample.dcm -threshold 90% \
-morphology HMT '5x5:0,0,0,0,0 0,1,1,1,1 0,1,-,-,- 0,1,-,-,- 0,1,-,-,-' result.png
现在我们还想寻找右上角、左下角和右下角,所以我们需要旋转图案,这可以通过添加
> 标志方便地由
ImageMagick 完成。
convert sample.dcm -threshold 90% \
-morphology HMT '5x5>:0,0,0,0,0 0,1,1,1,1 0,1,-,-,- 0,1,-,-,- 0,1,-,-,-' result.png

希望您现在能看到标志角落的点,这样我们就可以请求 ImageMagick 剪裁所有多余的黑色图像,只留下白色的点,并告诉我们边界框:
cconvert sample.dcm -threshold 90% \
-morphology HMT '5x5>:0,0,0,0,0 0,1,1,1,1 0,1,-,-,- 0,1,-,-,- 0,1,-,-,-' -format %@ info:
308x198+1822+427
因此,如果我现在在这些坐标周围画一个红色框,你就可以看到标签被检测到的位置 - 当然,在实践中,我会画一个黑色框来覆盖它,但我正在解释这个想法:
convert sample.dcm -fill "rgba(255,0,0,0.5)" -draw "rectangle 1822,427 2130,625" result.png
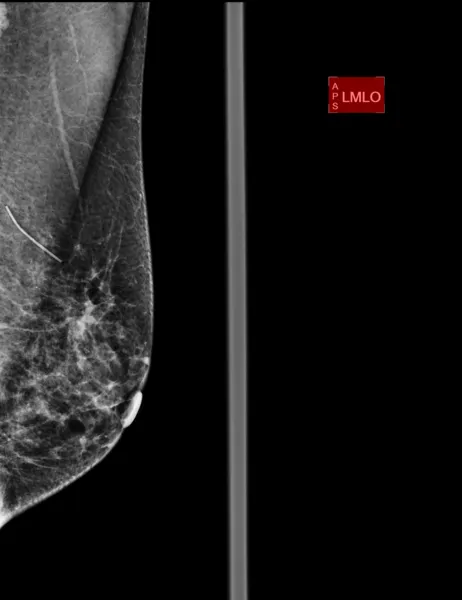
如果您想让脚本自动完成这个任务,我建议使用以下代码,并将其保存为
HideMarker:
#!/bin/bash
input="$1"
output="$2"
IFS="x+" read w h x1 y1 < <(convert "$input" -threshold 90% -morphology HMT '5x5>:0,0,0,0,0 0,1,1,1,1 0,1,-,-,- 0,1,-,-,- 0,1,-,-,-' -format %@ info:)
((x1=x1-1))
((y1=y1-1))
((x2=x1+w+1))
((y2=y1+h+1))
convert "$input" -fill black -draw "rectangle $x1,$y1 $x2,$y2" "$output"
然后您需要执行以下操作使其可执行:
chmod +x HideMarker
然后像这样运行:
./HideMarker someImage.dcm result.png
 。
。