一种可能的解决方案是使用
scipy.interpolate.RectBivariateSpline。在下面的代码中,
x_0和
y_0是数据中特征的坐标(即您示例中高斯分布的中心位置)需要映射到由
coords给出的坐标。这种方法有几个优点:
如果您需要将相同的对象“放置”到输出frame中的多个位置,则只需计算样条曲线一次(但要评估多次)。
如果您实际上需要计算模型在像素上的积分通量,则可以使用scipy.interpolate.RectBivariateSpline的integral method。
使用样条插值重新采样:
from scipy.interpolate import RectBivariateSpline
x = np.arange(data.shape[1], dtype=np.float)
y = np.arange(data.shape[0], dtype=np.float)
kx = 3; ky = 3;
spline = RectBivariateSpline(
x, y, data.T, kx=kx, ky=ky, s=0
)
x_0 = (data.shape[1] - 1.0) / 2.0
y_0 = (data.shape[0] - 1.0) / 2.0
yg, xg = np.indices(frame.shape, dtype=np.float64)
xg += x_0 - coords[0]
yg += y_0 - coords[1]
resampled_data = spline.ev(xg, yg)
extrapol = (((xg < -0.5) | (xg >= data.shape[1] - 0.5)) |
((yg < -0.5) | (yg >= data.shape[0] - 0.5)))
resampled_data[extrapol] = 0
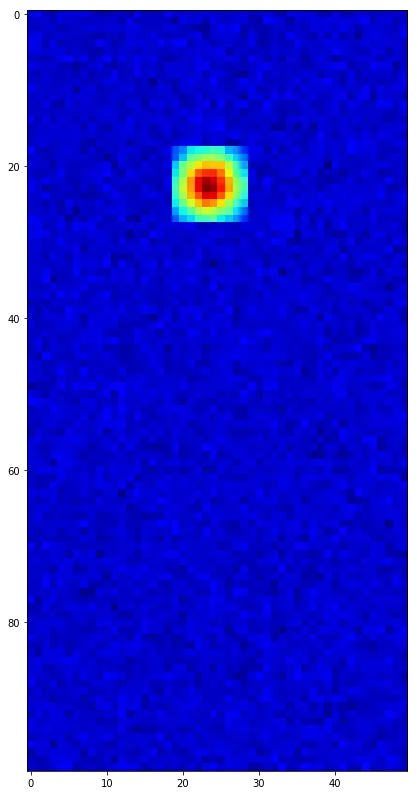
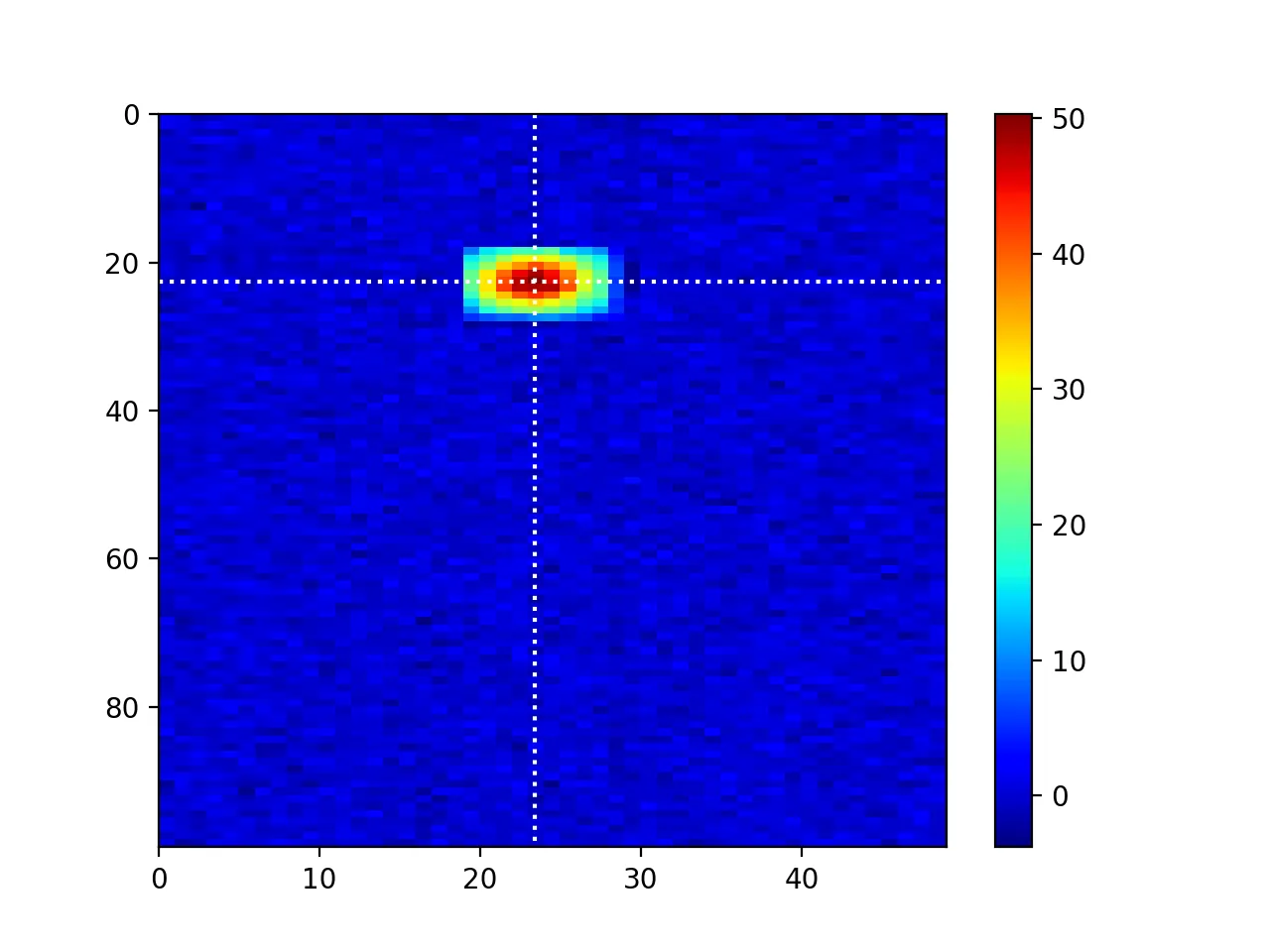
现在绘制
frame和重采样的
data:
plt.figure(figsize=(14, 14))
plt.imshow(frame+resampled_data, cmap=plt.cm.jet,
origin='upper', interpolation='none', aspect='equal')
plt.show()
如果你也希望允许比例变化,那么请将上面计算xg和yg的代码用以下代码替换:
coords = 20, 80
zoom_x = 2
zoom_y = 3
yg, xg = np.indices(frame.shape, dtype=np.float64)
xg = (xg - coords[0]) / zoom_x + x_0
yg = (yg - coords[1]) / zoom_y + y_0
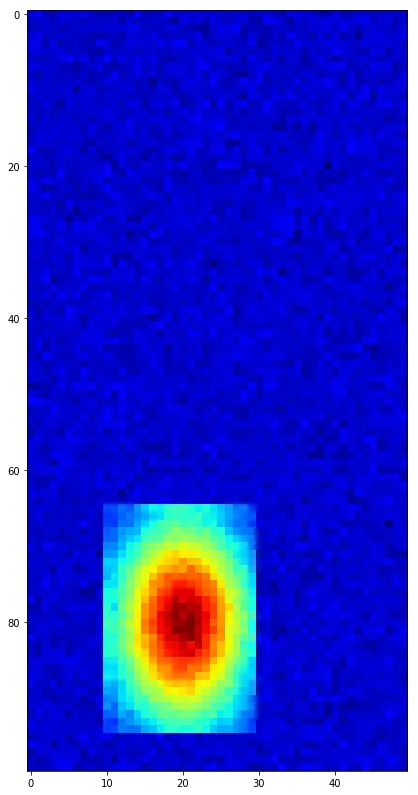
根据您的示例,最有可能这正是您实际想要的。具体而言,data中像素的坐标以0.222(2)距离单位“间隔”。因此,对于您特定的示例(无论是意外还是故意),您实际上具有0.222(2)的缩放因子。在这种情况下,您的data图像将在输出帧中缩小到几乎2个像素。
与@Chiel答案的比较
在下面的图像中,我比较了我的方法(左侧)、@Chiel的方法(中间)和差异(右侧面板)的结果:
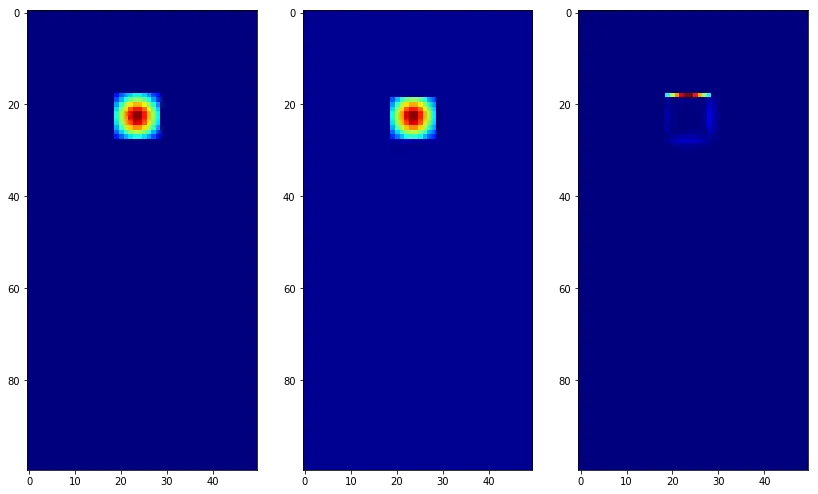
基本上,这两种方法非常相似,甚至可能使用相同的算法(我没有查看
shift的代码,但根据描述 - 它也使用样条曲线)。从比较图像中可以看出,最大的差异在于边缘,并且由于我不知道原因,
shift似乎会过早地截断移动后的图像。
我认为最大的区别在于我的方法允许像素比例的更改,它还允许重复使用相同的插值器将原始图像放置在输出帧的不同位置。
@Chiel的方法有点简单,但(我不喜欢的是)它需要创建一个更大的数组(
data_large),将原始图像放置在角落里。




frame的坐标是什么?所以你只想用data替换frame的一部分? - anishtain4frame的坐标只是索引,而不是替换。我想要添加它。 - Joe Flipshift可以完成。如果不是,则另一个答案是最通用和最好的。 - Chieldata图像中的像素“坐标”(x和y)间隔为0.222(2)个单位(“像素比例尺”)-请参见np.linspace(-1,1,10),因此,如果映射到输出frame网格上(假设间距为1个像素),则将导致data图像在放置到输出frame图像中时缩小至仅2个像素大小。 但是,您的示例图像没有显示这种缩放变换。因此,我的问题是:linspace用于生成“真实”的坐标网格还是仅用于为高斯和data像素的像素网格创建网格? - AGN Gazer