事实上有两个问题,其中一个比另一个更高级。
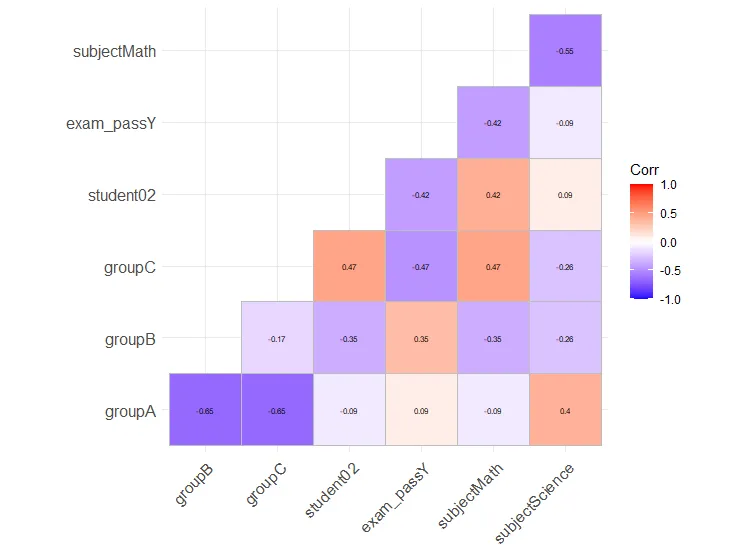
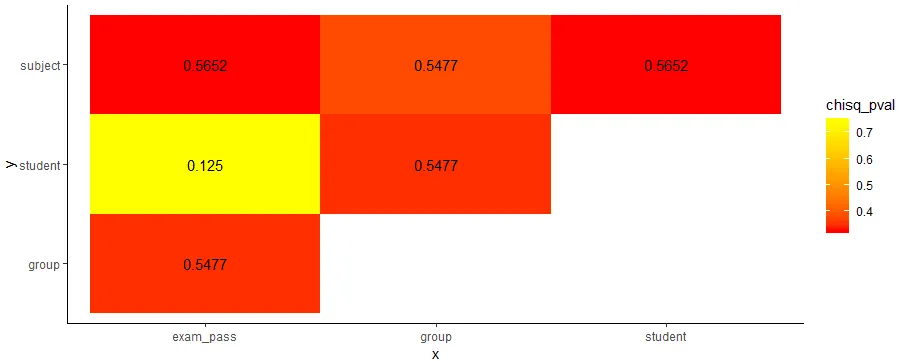
Q1:我正在寻找一种类似于corrplot()的方法,但可以处理因子数据。
我最初尝试使用chisq.test()然后计算p值和Cramer's V作为相关性,但是由于列太多而无法解决。
所以,有人能告诉我是否有一种快速创建“corrplot”的方法,使每个单元格都包含Cramer's V的值,而颜色是由p值呈现的。或者任何其他类似的图表。
关于Cramer's V,假设tbl是一个二维因子数据框。
chi2 <- chisq.test(tbl, correct=F)
Cramer_V <- sqrt(chi2$/nrow(tbl))
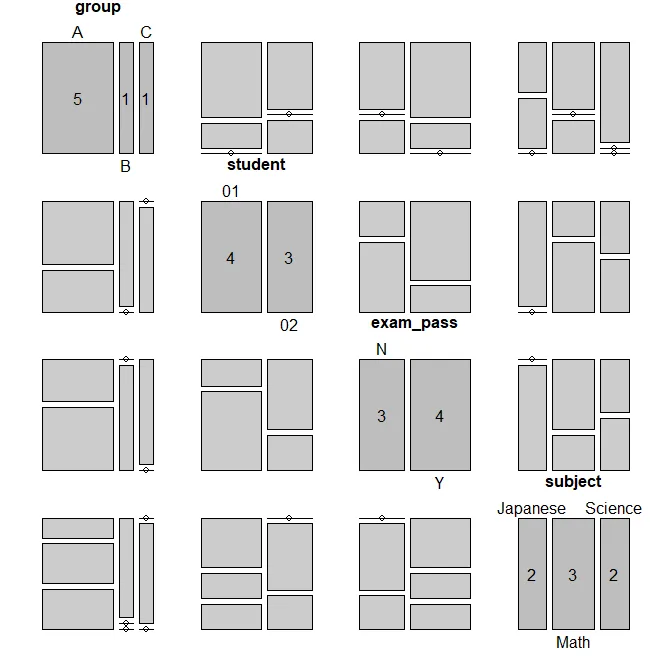
我准备了一个包含因子的测试数据框:
df <- data.frame(
group = c('A', 'A', 'A', 'A', 'A', 'B', 'C'),
student = c('01', '01', '01', '02', '02', '01', '02'),
exam_pass = c('Y', 'N', 'Y', 'N', 'Y', 'Y', 'N'),
subject = c('Math', 'Science', 'Japanese', 'Math', 'Science', 'Japanese', 'Math')
)
问题2:我希望能够在一个混合类型的数据框上计算相关性/关联矩阵,例如:
df <- data.frame(
group = c('A', 'A', 'A', 'A', 'A', 'B', 'C'),
student = c('01', '01', '01', '02', '02', '01', '02'),
exam_pass = c('Y', 'N', 'Y', 'N', 'Y', 'Y', 'N'),
subject = c('Math', 'Science', 'Japanese', 'Math', 'Science', 'Japanese', 'Math')
)
df$group <- factor(df$group, levels = c('A', 'B', 'C'), ordered = T)
df$student <- as.integer(df$student)