我在SAS LIFEREG中有一个加速失效时间模型,我想绘制它。由于 SAS 在绘图方面极其糟糕,我想在 R 中重新生成曲线的数据并在那里绘制它们。SAS 输出了一个比例尺(在指数分布固定为1的情况下)、一个截距和一个回归系数,用于暴露或未暴露人群。
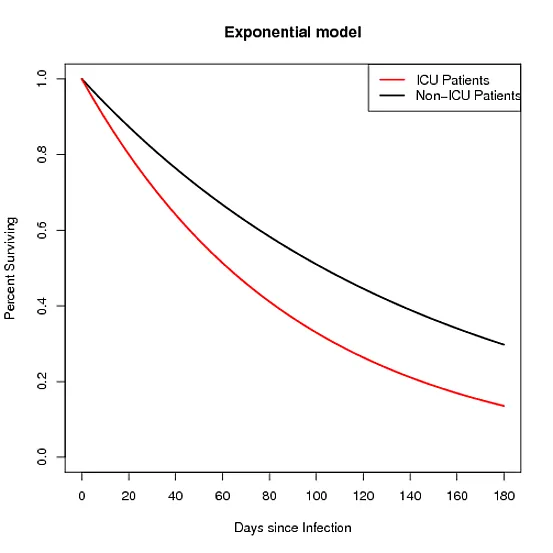
有两条曲线,一条是暴露人群的,另一条是未暴露人群的。其中一个模型是指数分布,我已经这样产生了数据和图形:
有两条曲线,一条是暴露人群的,另一条是未暴露人群的。其中一个模型是指数分布,我已经这样产生了数据和图形:
intercept <- 5.00
effect<- -0.500
data<- data.frame(time=seq(0:180)-1)
data$s_unexposed <- apply(data,1,function(row) exp(-(exp(-intercept))*row[1]))
data$s_exposed <- apply(data,1,function(row) exp(-(exp(-(intercept+effect))*row[1])))
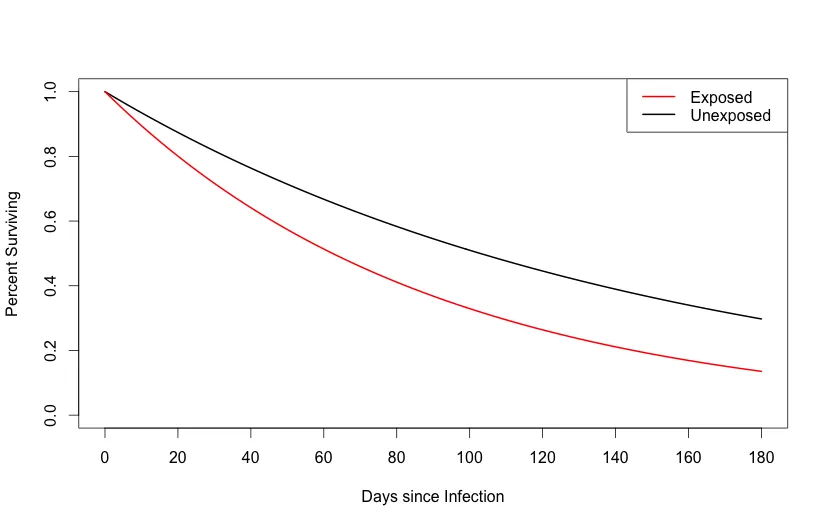
plot(data$time,data$s_unexposed, type="l", ylim=c(0,1) ,xaxt='n',
xlab="Days since Infection", ylab="Percent Surviving", lwd=2)
axis(1, at=c(0, 20, 40, 60, 80, 100, 120, 140, 160, 180))
lines(data$time,data$s_exposed, col="red",lwd=2)
legend("topright", c("ICU Patients", "Non-ICU Patients"), lwd=2, col=c("red","black") )
这给我带来了以下结果:
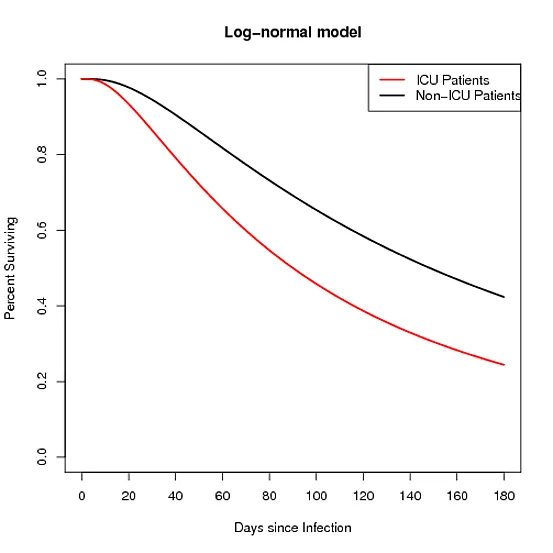
虽然不是最美丽的图表,但我并不太了解如何使用ggplot2来美化它。更重要的是,我有第二组数据,其来源于对数正态分布,而不是指数分布,我的尝试生成该数据已经彻底失败了 - 对于正态分布的累积分布函数等的融合将其超出了我的R技能范围。
有没有人能够指导我朝正确的方向前进,使用相同的数字和1的比例参数?