我正在尝试使用Eigen在C++中从数据集计算2个主要的主成分。我目前的做法是将数据标准化为
以下是我如何实现它的主要代码(其中
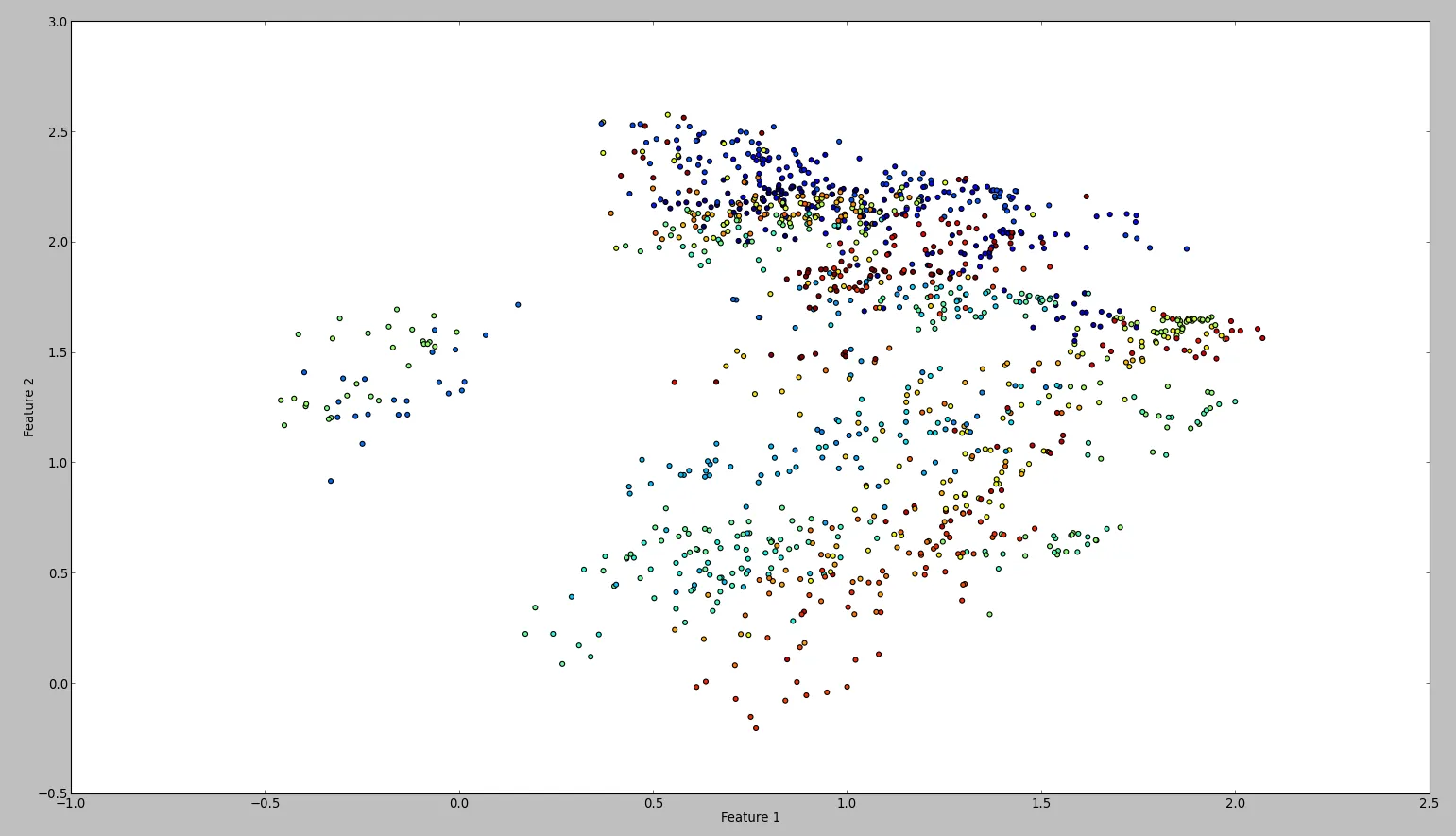
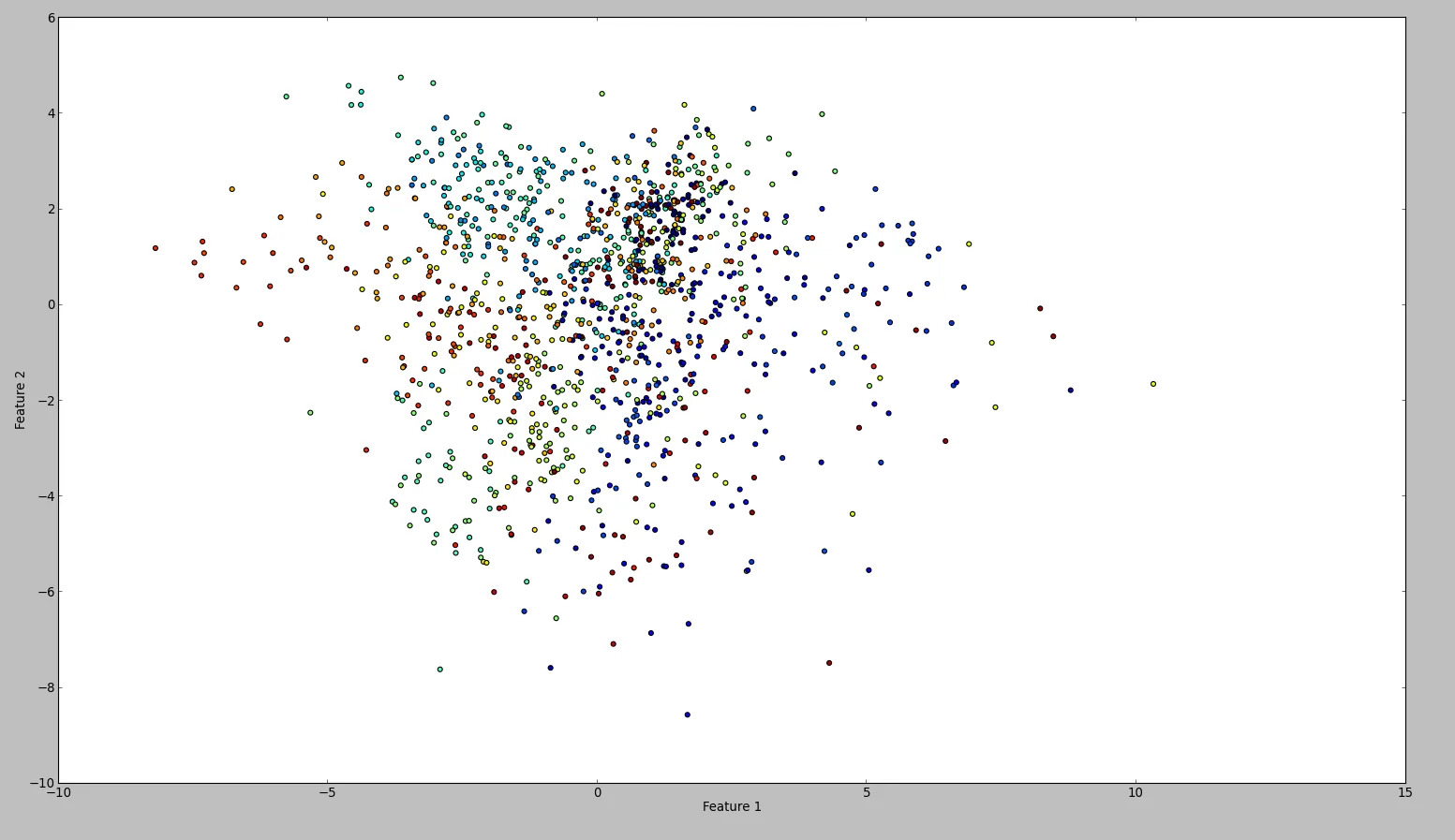
这段代码的结果大致如下: 为了确认我的结果,我尝试使用WEKA进行相同的操作。所以我按照这个顺序使用了归一化和居中过滤器,然后是主成分过滤器,并保存并绘制输出。结果如下: 从技术上讲,我应该做同样的事情,但结果却非常不同。有人能看出我是否犯了错误吗?
[0,1]之间,并使其居中。之后,我计算协方差矩阵并对其进行特征值分解。我知道SVD更快,但是我对计算出的主成分感到困惑。以下是我如何实现它的主要代码(其中
traindata是我的MxN大小的输入矩阵):Eigen::VectorXf normalize(Eigen::VectorXf vec) {
for (int i = 0; i < vec.size(); i++) { // normalize each feature.
vec[i] = (vec[i] - minCoeffs[i]) / scalingFactors[i];
}
return vec;
}
// Calculate normalization coefficients (globals of type Eigen::VectorXf).
maxCoeffs = traindata.colwise().maxCoeff();
minCoeffs = traindata.colwise().minCoeff();
scalingFactors = maxCoeffs - minCoeffs;
// For each datapoint.
for (int i = 0; i < traindata.rows(); i++) { // Normalize each datapoint.
traindata.row(i) = normalize(traindata.row(i));
}
// Mean centering data.
Eigen::VectorXf featureMeans = traindata.colwise().mean();
Eigen::MatrixXf centered = traindata.rowwise() - featureMeans;
// Compute the covariance matrix.
Eigen::MatrixXf cov = centered.adjoint() * centered;
cov = cov / (traindata.rows() - 1);
Eigen::SelfAdjointEigenSolver<Eigen::MatrixXf> eig(cov);
// Normalize eigenvalues to make them represent percentages.
Eigen::VectorXf normalizedEigenValues = eig.eigenvalues() / eig.eigenvalues().sum();
// Get the two major eigenvectors and omit the others.
Eigen::MatrixXf evecs = eig.eigenvectors();
Eigen::MatrixXf pcaTransform = evecs.rightCols(2);
// Map the dataset in the new two dimensional space.
traindata = traindata * pcaTransform;
这段代码的结果大致如下: 为了确认我的结果,我尝试使用WEKA进行相同的操作。所以我按照这个顺序使用了归一化和居中过滤器,然后是主成分过滤器,并保存并绘制输出。结果如下: 从技术上讲,我应该做同样的事情,但结果却非常不同。有人能看出我是否犯了错误吗?