我正在尝试缩短一个执行两个矩阵系列计算的函数时间。在搜索过程中,我听说过numpy,但我真的不知道如何将其应用于我的问题。此外,我认为使我的函数变慢的原因之一是有许多点运算符(我在这个this page中听说过)。
这个数学问题对应于二次分配问题的分解:
我尝试将
我该如何减少时间?
编辑1
回答eickenberg的评论:
一个最小的工作示例:
现在我认为更好的选择是使用NumPy。我试过Matrix(),但没有改善性能。
找到的最佳解决方案
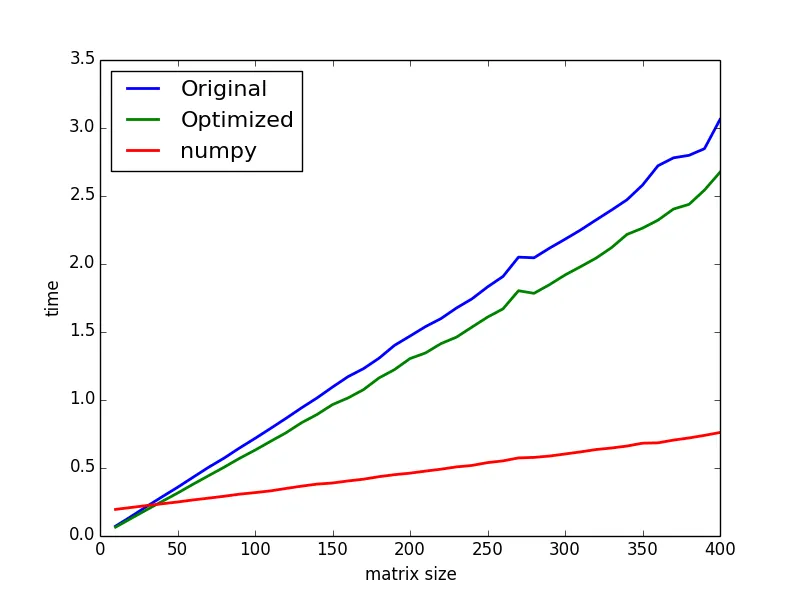
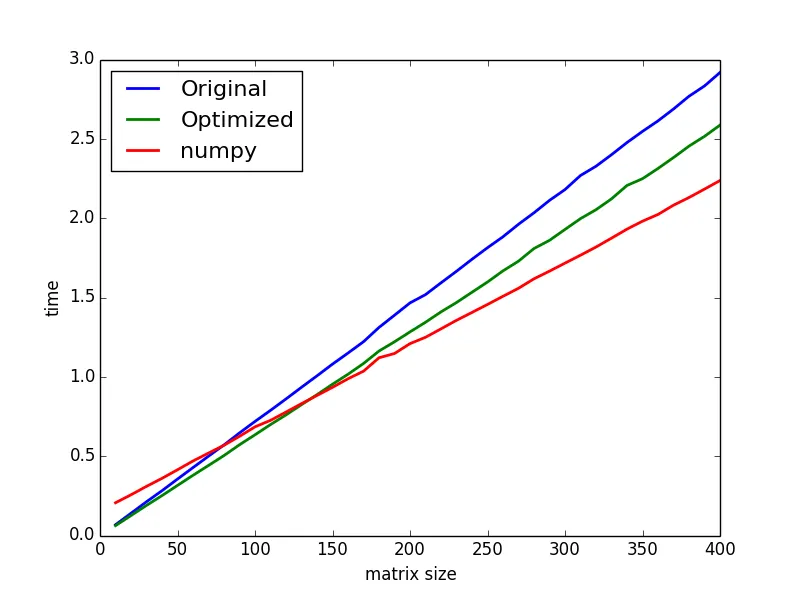
嗯,最终我能够通过结合@TooTone的解决方案并将索引存储在集合中来减少时间,以避免if语句。时间从大约18秒降至8秒。这是代码:
为了进一步缩短时间,我认为最好的方法是编写一个模块。
这个数学问题对应于二次分配问题的分解:
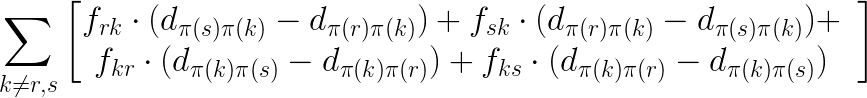
我的代码是:
delta = 0
for k in xrange(self._tam):
if k != r and k != s:
delta +=
self._data.stream_matrix[r][k] \
* (self._data.distance_matrix[sol[s]][sol[k]] - self._data.distance_matrix[sol[r]][sol[k]]) + \
self._data.stream_matrix[s][k] \
* (self._data.distance_matrix[sol[r]][sol[k]] - self._data.distance_matrix[sol[s]][sol[k]]) + \
self._data.stream_matrix[k][r] \
* (self._data.distance_matrix[sol[k]][sol[s]] - self._data.distance_matrix[sol[k]][sol[r]]) + \
self._data.stream_matrix[k][s] \
* (self._data.distance_matrix[sol[k]][sol[r]] - self._data.distance_matrix[sol[k]][sol[s]])
return delta
在处理大小为20的问题时(20x20的矩阵),需要大约20秒钟,瓶颈在于这个函数。
ncalls tottime percall cumtime percall filename:lineno(function)
303878 15.712 0.000 15.712 0.000 Heuristic.py:66(deltaC)
我尝试将
map应用于for循环,但由于循环体不是函数调用,因此不可能实现。我该如何减少时间?
编辑1
回答eickenberg的评论:
sol是一个排列,例如[1,2,3,4]。当我生成相邻解时调用该函数,因此[1,2,3,4]的相邻值是[2,1,3,4]。我只更改原始排列中的两个位置,然后调用deltaC,它计算交换位置r、s的解的分解(在上面的示例中,r、s = 0,1)。这个排列是为了避免计算相邻解的整个成本。我想我可以将sol[k,r,s]的值存储在本地变量中,以避免在每次迭代中查找其值。我不知道这是否是您在评论中询问的内容。
编辑2一个最小的工作示例:
import random
distance_matrix = [[0, 12, 6, 4], [12, 0, 6, 8], [6, 6, 0, 7], [4, 8, 7, 0]]
stream_matrix = [[0, 3, 8, 3], [3, 0, 2, 4], [8, 2, 0, 5], [3, 4, 5, 0]]
def deltaC(r, s, S=None):
'''
Difference between C with values i and j swapped
'''
S = [0,1,2,3]
if S is not None:
sol = S
else:
sol = S
delta = 0
sol_r, sol_s = sol[r], sol[s]
for k in xrange(4):
if k != r and k != s:
delta += (stream_matrix[r][k] \
* (distance_matrix[sol_s][sol[k]] - distance_matrix[sol_r][sol[k]]) + \
stream_matrix[s][k] \
* (distance_matrix[sol_r][sol[k]] - distance_matrix[sol_s][sol[k]]) + \
stream_matrix[k][r] \
* (distance_matrix[sol[k]][sol_s] - distance_matrix[sol[k]][sol_r]) + \
stream_matrix[k][s] \
* (distance_matrix[sol[k]][sol_r] - distance_matrix[sol[k]][sol_s]))
return delta
for _ in xrange(303878):
d = deltaC(random.randint(0,3), random.randint(0,3))
print d
现在我认为更好的选择是使用NumPy。我试过Matrix(),但没有改善性能。
找到的最佳解决方案
嗯,最终我能够通过结合@TooTone的解决方案并将索引存储在集合中来减少时间,以避免if语句。时间从大约18秒降至8秒。这是代码:
def deltaC(self, r, s, sol=None):
delta = 0
sol = self.S if sol is None else self.S
sol_r, sol_s = sol[r], sol[s]
stream_matrix = self._data.stream_matrix
distance_matrix = self._data.distance_matrix
indexes = set(xrange(self._tam)) - set([r, s])
for k in indexes:
sol_k = sol[k]
delta += \
(stream_matrix[r][k] - stream_matrix[s][k]) \
* (distance_matrix[sol_s][sol_k] - distance_matrix[sol_r][sol_k]) \
+ \
(stream_matrix[k][r] - stream_matrix[k][s]) \
* (distance_matrix[sol_k][sol_s] - distance_matrix[sol_k][sol_r])
return delta
为了进一步缩短时间,我认为最好的方法是编写一个模块。
s,如sol[s],即使它保持不变。在尝试呈现解决方案之前,如果您能告诉我们是否必须针对所有r, s执行此操作以及sol是否为索引的固定排列,则会非常好。如果向量化无法奏效(虽然应该可以),则可以尝试编译数值表达式,例如使用numexpr,但我建议将其留到更后面。 - eickenbergself._data.stream_matrix和self._data.distance_matrix是numpy矩阵类,那么效率会更高吗? - Alejandro Alcaldedelta的值)。矩阵不必像20x20那样大,您只需要为矩阵添加示例数据,对于sol,请将对self._tam的引用替换为常量等。 - TooTone