import numpy as np
import pandas as pd
from scipy.optimize import curve_fit
import matplotlib.pyplot as plt
%matplotlib inline
# power law function
def func_powerlaw(x,a,b,c):
return a*(x**b)+c
test_X = [1.0,2,3,4,5,6,7,8,9,10]
test_Y =[3.0,1.5,1.2222222222222223,1.125,1.08,1.0555555555555556,1.0408163265306123,1.03125, 1.0246913580246915,1.02]
predict_Y = []
for x in test_X:
predict_Y.append(2*x**-2+1)
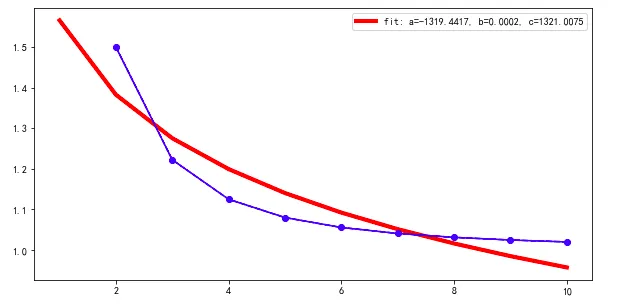
如果我使用默认的初始猜测值,即 p0 = [1,1,1] 进行对齐
popt, pcov = curve_fit(func_powerlaw, test_X[1:], test_Y[1:], maxfev=2000)
plt.figure(figsize=(10, 5))
plt.plot(test_X, func_powerlaw(test_X, *popt),'r',linewidth=4, label='fit: a=%.4f, b=%.4f, c=%.4f' % tuple(popt))
plt.plot(test_X[1:], test_Y[1:], '--bo')
plt.plot(test_X[1:], predict_Y[1:], '-b')
plt.legend()
plt.show()
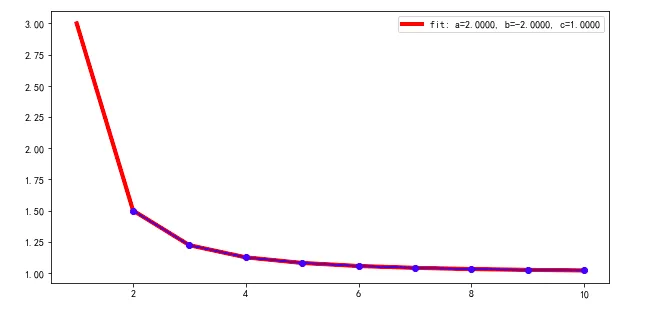
如果我将初始猜测值更改为p0 = [0.5,0.5,0.5]
popt, pcov = curve_fit(func_powerlaw, test_X[1:], test_Y[1:], p0=np.asarray([0.5,0.5,0.5]), maxfev=2000)
我可以得到最佳拟合
 ---------------------2018年10月7日更新-------------------------------------------------------------------------------------------------------------------------
---------------------2018年10月7日更新-------------------------------------------------------------------------------------------------------------------------由于我需要运行数千甚至数百万个Power Law函数,使用@James Phillips的方法太昂贵了。除了curve_fit之外,什么方法是适当的?例如sklearn,np.linalg.lstsq等。