我正在使用lme4包运行glmer logit模型。我对各种二元和三元交互作用效应及其解释感兴趣。为简化问题,我只关心固定效应系数。
我设法编写了一段代码来计算并绘制这些效应的logit比例,但我在将它们转换为预测概率比例时遇到了困难。最终,我希望复制“effects”包的输出结果。
该示例依赖于UCLA癌症患者数据。
尽管我不确定是否应该担心这个警告,但我使用估计值来绘制所关注的交互作用的平均边际效应。首先,我准备将数据集馈送到
我认为现在的OY比例是以logit为单位度量的,但为了理解它,我希望将其转换为预测概率。根据wikipedia,类似于
即使我可以使用
修复这个问题需要调整估计过程,但目前我想避免这样做。此外,我也很好奇
我设法编写了一段代码来计算并绘制这些效应的logit比例,但我在将它们转换为预测概率比例时遇到了困难。最终,我希望复制“effects”包的输出结果。
该示例依赖于UCLA癌症患者数据。
library(lme4)
library(ggplot2)
library(plyr)
getmode <- function(v) {
uniqv <- unique(v)
uniqv[which.max(tabulate(match(v, uniqv)))]
}
facmin <- function(n) {
min(as.numeric(levels(n)))
}
facmax <- function(x) {
max(as.numeric(levels(x)))
}
hdp <- read.csv("http://www.ats.ucla.edu/stat/data/hdp.csv")
head(hdp)
hdp <- hdp[complete.cases(hdp),]
hdp <- within(hdp, {
Married <- factor(Married, levels = 0:1, labels = c("no", "yes"))
DID <- factor(DID)
HID <- factor(HID)
CancerStage <- revalue(hdp$CancerStage, c("I"="1", "II"="2", "III"="3", "IV"="4"))
})
直到这里都是关于数据管理、函数和我需要的包。
m <- glmer(remission ~ CancerStage*LengthofStay + Experience +
(1 | DID), data = hdp, family = binomial(link="logit"))
summary(m)
这是模型。它需要一分钟,并收敛并显示以下警告:
Warning message:
In checkConv(attr(opt, "derivs"), opt$par, ctrl = control$checkConv, :
Model failed to converge with max|grad| = 0.0417259 (tol = 0.001, component 1)
尽管我不确定是否应该担心这个警告,但我使用估计值来绘制所关注的交互作用的平均边际效应。首先,我准备将数据集馈送到
predict函数中,然后使用固定效应参数计算边际效应以及置信区间。newdat <- expand.grid(
remission = getmode(hdp$remission),
CancerStage = as.factor(seq(facmin(hdp$CancerStage), facmax(hdp$CancerStage),1)),
LengthofStay = seq(min(hdp$LengthofStay, na.rm=T),max(hdp$LengthofStay, na.rm=T),1),
Experience = mean(hdp$Experience, na.rm=T))
mm <- model.matrix(terms(m), newdat)
newdat$remission <- predict(m, newdat, re.form = NA)
pvar1 <- diag(mm %*% tcrossprod(vcov(m), mm))
cmult <- 1.96
## lower and upper CI
newdat <- data.frame(
newdat, plo = newdat$remission - cmult*sqrt(pvar1),
phi = newdat$remission + cmult*sqrt(pvar1))
我相信这些是在logit比例尺上的正确估计,但也许我错了。无论如何,这是图表:
plot_remission <- ggplot(newdat, aes(LengthofStay,
fill=factor(CancerStage), color=factor(CancerStage))) +
geom_ribbon(aes(ymin = plo, ymax = phi), colour=NA, alpha=0.2) +
geom_line(aes(y = remission), size=1.2) +
xlab("Length of Stay") + xlim(c(2, 10)) +
ylab("Probability of Remission") + ylim(c(0.0, 0.5)) +
labs(colour="Cancer Stage", fill="Cancer Stage") +
theme_minimal()
plot_remission
我认为现在的OY比例是以logit为单位度量的,但为了理解它,我希望将其转换为预测概率。根据wikipedia,类似于
exp(value)/(exp(value)+1)这样的公式可以得到预测概率。虽然我可以使用newdat$remission <- exp(newdat$remission)/(exp(newdat$remission)+1),但我不确定应该如何处理置信区间。
最终,我想得到与effects包生成的相同图形。即:eff.m <- effect("CancerStage*LengthofStay", m, KR=T)
eff.m <- as.data.frame(eff.m)
plot_remission2 <- ggplot(eff.m, aes(LengthofStay,
fill=factor(CancerStage), color=factor(CancerStage))) +
geom_ribbon(aes(ymin = lower, ymax = upper), colour=NA, alpha=0.2) +
geom_line(aes(y = fit), size=1.2) +
xlab("Length of Stay") + xlim(c(2, 10)) +
ylab("Probability of Remission") + ylim(c(0.0, 0.5)) +
labs(colour="Cancer Stage", fill="Cancer Stage") +
theme_minimal()
plot_remission2
即使我可以使用
effects包,但是它不幸地无法与我必须运行的许多模型编译。Error in model.matrix(mod2) %*% mod2$coefficients :
non-conformable arguments
In addition: Warning message:
In vcov.merMod(mod) :
variance-covariance matrix computed from finite-difference Hessian is
not positive definite or contains NA values: falling back to var-cov estimated from RX
修复这个问题需要调整估计过程,但目前我想避免这样做。此外,我也很好奇
effects 在这里实际上是做什么。
如果有任何关于如何调整我的初始语法以获得预测概率的建议,我将不胜感激!
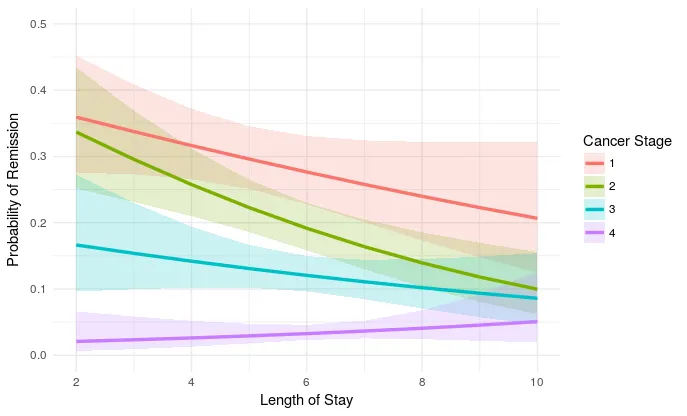
ggplot(newdat, aes(LengthofStay, fill=factor(CancerStage), color=factor(CancerStage))) + geom_ribbon(aes(ymin=plo, ymax=phi), colour=NA, alpha=0.2) + geom_line(aes(y = remission), size=1.2) + xlab("住院时间") + ylab("缓解概率") + labs(colour="癌症分期", fill="癌症分期") + theme_minimal()- eipi10getAnywhere(Effect.default)来获取由Effects包运行的代码。 - Ian Wesley