方法1a
可以使用np.lexsort、np.unique和np.ufunc.reduceat来完成:
A = A[np.lexsort((A[:, 0], A[:, 1]))]
_, idx = np.unique(A[:,:2], return_index = True, axis=0)
output = np.maximum.reduceat(A, idx)
方法1b
我们也可以以稍微高效的方式提高reduceat的速度:
A = A[np.lexsort((A[:, 0], A[:, 1]))]
u, idx = np.unique(A[:, :2], return_index = True, axis=0)
return np.c_[u, np.maximum.reduceat(A[:,2], idx)]
方法二
如果您想要一个更简单的方法,您也可以使用 numpy_indexed,它是基于 numpy 编写的,并允许用更少的脚本解决分组问题:
import numpy_indexed as npi
_, idx = npi.group_by(A[:, :2]).argmax(A[:, 2])
output = A[idx]
注意,它的性能优于以前的方法,这表明它可以进一步优化。
方法3
与numpy相比,pandas的某些方法更快。看起来你在使用方法上很幸运:
import pandas as pd
df = pd.DataFrame(A)
return df.loc[df.groupby([0,1])[2].idxmax()].values
输出
所有的输出结果都是:np.array([[0. 0. 2.], [1. 1. 3.], [1. 2. 1.]]),除了使用npi方法的情况,结果为np.array([[0. 0. 2.], [1. 2. 1.], [1. 1. 3.]])
更新
如果在降维后的平坦数组上执行相同的算法,可以进一步优化。使用numexpr包可以实现极速运行。新方法的名称为:approach1a_on1D,approach1b_on1D,approach2_on1D。
import numexpr as ne
def reduct_dims(cubes):
m0, m1 = np.min(cubes[:,:2], axis=0)
M0 = np.max(cubes[:,0], axis=0)
s0 = M0 - m0 + 1
d = {'c0':cubes[:,0],'c1':cubes[:,1],'c2':cubes[:,2],
's0':s0,'m0':m0, 'm1':m1}
return ne.evaluate('c0-m0+(c1-m1)*s0', d)
def approach1a(A):
A = A[np.lexsort((A[:, 0], A[:, 1]))]
_, idx = np.unique(A[:, :2], return_index = True, axis=0)
return np.maximum.reduceat(A, idx)
def approach1b(A):
A = A[np.lexsort((A[:, 0], A[:, 1]))]
u, idx = np.unique(A[:, :2], return_index = True, axis=0)
return np.c_[u, np.maximum.reduceat(A[:,2], idx)]
def approach2(A):
_, idx = npi.group_by(A[:, :2]).argmax(A[:, 2])
return A[idx]
def approach3(A):
df = pd.DataFrame(A)
return df.loc[df.groupby([0,1])[2].idxmax()].values
def approach1a_on1D(A):
A_as_1D = reduct_dims(A)
argidx = np.argsort(A_as_1D)
A, A_as_1D = A[argidx], A_as_1D[argidx]
_, idx = np.unique(A_as_1D, return_index = True)
return np.maximum.reduceat(A, idx)
def approach1b_on1D(A):
A_as_1D = reduct_dims(A)
argidx = np.argsort(A_as_1D)
A, A_as_1D = A[argidx], A_as_1D[argidx]
_, idx = np.unique(A_as_1D, return_index = True)
return np.c_[A[:,:2][idx], np.maximum.reduceat(A[:,2], idx)]
def approach2_on1D(A):
A_as_1D = reduct_dims(A)
_, idx = npi.group_by(A_as_1D).argmax(A[:,2])
return A[idx]
%timeit reduct_dims(cubes)
160 ms ± 7.44 ms per loop (mean ± std. dev. of 7 runs, 10
loops each)
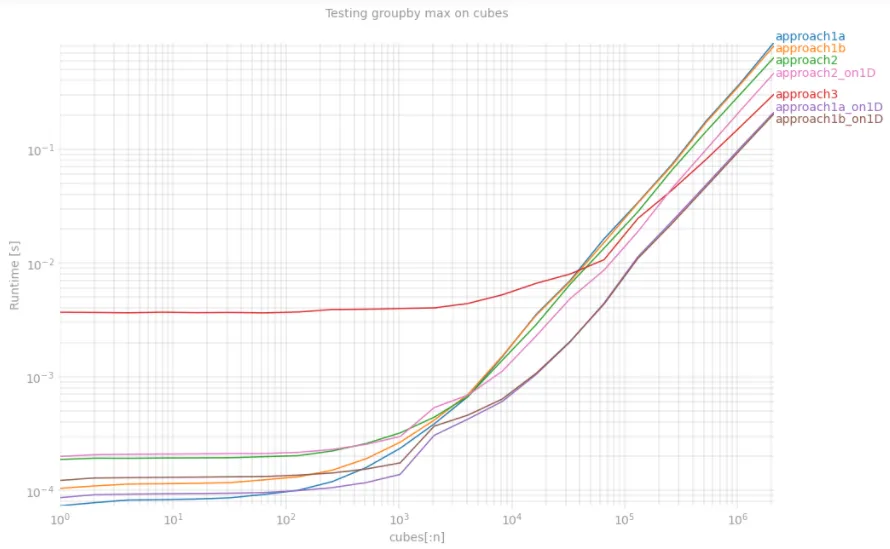
使用perfplot分析性能
我对从激光雷达获取的实际点云数据进行了测试,该数据以厘米为单位给出3D值(约20M个点,其中1M个是不同的)。最快的版本在2秒内运行。让我们看看在perfplot上的结果:
import tensorflow as tf
import perfplot
import matplotlib.pyplot as plt
from time import time
path = tf.keras.utils.get_file('cubes.npz', 'https://github.com/loijord/lidar_home/raw/master/cubes.npz')
cubes = np.load(path)['array'].astype(np.int64) // 50
t = time()
fig = plt.figure(figsize=(15, 10))
plt.grid(True, which="both")
out = perfplot.bench(
setup = lambda x: cubes[:x],
kernels = [approach1a, approach1b, approach2, approach3, approach1a_on1D, approach1b_on1D, approach2_on1D],
n_range = [2 ** k for k in range(22)],
xlabel = 'cubes[:n]',
title = 'Testing groupby max on cubes',
show_progress = False,
equality_check = False)
out.show()
print('Overall testing time:', time() -t)