情况 k = 2:np.triu_indices
我已经使用许多变化的上述函数和perfplot测试了情况 k = 2。毫无疑问,胜利者是np.triu_indices,现在我看到,即使对于像igraph.EdgeList这样的奇特数据类型,使用np.dtype([('', np.intp)] * 2)数据结构也可以大大提高性能。
from itertools import combinations, chain
from scipy.special import comb
import igraph as ig
import networkx as nx
def _combs(n):
return np.array(list(combinations(range(n),2)))
def _combs_fromiter(n):
indices = np.arange(n)
dt = np.dtype([('', np.intp)]*2)
indices = np.fromiter(combinations(indices, 2), dt)
indices = indices.view(np.intp).reshape(-1, 2)
return indices
def _combs_fromiterplus(n):
dt = np.dtype([('', np.intp)]*2)
indices = np.fromiter(combinations(range(n), 2), dt)
indices = indices.view(np.intp).reshape(-1, 2)
return indices
def _numpy(n):
return np.transpose(np.triu_indices(n,1))
def _igraph(n):
return np.array(ig.Graph(n).complementer(False).get_edgelist())
def _igraph_fromiter(n):
dt = np.dtype([('', np.intp)]*2)
indices = np.fromiter(ig.Graph(n).complementer(False).get_edgelist(), dt)
indices = indices.view(np.intp).reshape(-1, 2)
return indices
def _nx(n):
G = nx.Graph()
G.add_nodes_from(range(n))
return np.array(list(nx.complement(G).edges))
def _nx_fromiter(n):
G = nx.Graph()
G.add_nodes_from(range(n))
dt = np.dtype([('', np.intp)]*2)
indices = np.fromiter(nx.complement(G).edges, dt)
indices = indices.view(np.intp).reshape(-1, 2)
return indices
def _comb_index(n):
count = comb(n, 2, exact=True)
index = np.fromiter(chain.from_iterable(combinations(range(n), 2)),
int, count=count*2)
return index.reshape(-1, 2)
fig = plt.figure(figsize=(15, 10))
plt.grid(True, which="both")
out = perfplot.bench(
setup = lambda x: x,
kernels = [_numpy, _combs, _combs_fromiter, _combs_fromiterplus,
_comb_index, _igraph, _igraph_fromiter, _nx, _nx_fromiter],
n_range = [2 ** k for k in range(12)],
xlabel = 'combinations(n, 2)',
title = 'testing combinations',
show_progress = False,
equality_check = False)
out.show()
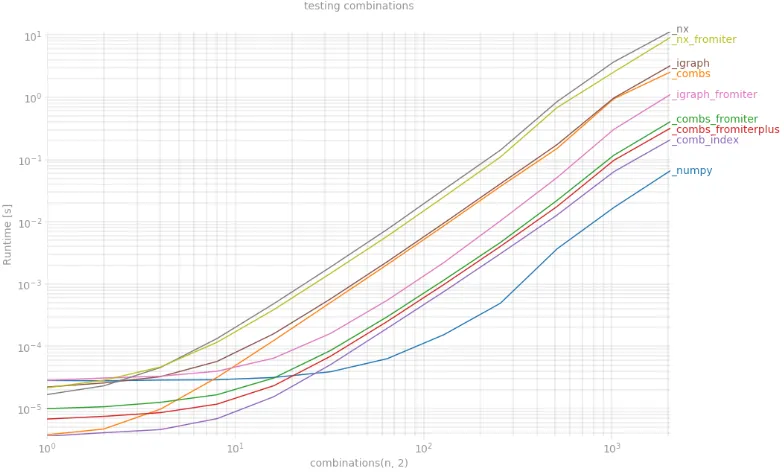
想知道为什么np.triu_indices不能扩展到更多维度吗?
情况2≤k≤4:triu_indices(此处实现) = 最多2倍的加速
np.triu_indices实际上可以成为k=3甚至k=4的优胜者,如果我们实现一个通用的方法。当前版本的方法相当于:
def triu_indices(n, k):
x = np.less.outer(np.arange(n), np.arange(-k+1, n-k+1))
return np.nonzero(x)
它构建了一个关系
x < y的矩阵表示,用于两个序列0,1,...,n-1,并找到它们不为零的单元格位置。对于3D情况,我们需要添加额外的维度并相交关系
x < y和
y < z。对于下一维,程序相同,但这会导致巨大的
内存超载,因为需要n^k个二进制单元格,而只有C(n,k)个达到True值。内存使用量和性能按O(n!)增长,因此该算法仅在k较小的情况下优于
itertools.combinations。实际上,这最好用于
k=2和
k=3的情况。
def C(n, k):
if k==0:
return np.array([])
if k==1:
return np.arange(1,n+1)
elif k==2:
return np.less.outer(np.arange(n), np.arange(n))
else:
x = C(n, k-1)
X = np.repeat(x[None, :, :], len(x), axis=0)
Y = np.repeat(x[:, :, None], len(x), axis=2)
return X&Y
def C_indices(n, k):
return np.transpose(np.nonzero(C(n,k)))
让我们使用perfplot进行结帐:
import matplotlib.pyplot as plt
import numpy as np
import perfplot
from itertools import chain, combinations
from scipy.special import comb
def C(n, k):
if k == 0:
return np.array([])
if k == 1:
return np.arange(1, n + 1)
elif k == 2:
return np.less.outer(np.arange(n), np.arange(n))
else:
x = C(n, k - 1)
X = np.repeat(x[None, :, :], len(x), axis=0)
Y = np.repeat(x[:, :, None], len(x), axis=2)
return X & Y
def C_indices(data):
n, k = data
return np.transpose(np.nonzero(C(n, k)))
def comb_index(data):
n, k = data
count = comb(n, k, exact=True)
index = np.fromiter(chain.from_iterable(combinations(range(n), k)),
int, count=count * k)
return index.reshape(-1, k)
def build_args(k):
return {'setup': lambda x: (x, k),
'kernels': [comb_index, C_indices],
'n_range': [2 ** x for x in range(2, {2: 10, 3:10, 4:7, 5:6}[k])],
'xlabel': f'N',
'title': f'test of case C(N,{k})',
'show_progress': True,
'equality_check': lambda x, y: np.array_equal(x, y)}
outs = [perfplot.bench(**build_args(n)) for n in (2, 3, 4, 5)]
fig = plt.figure(figsize=(20, 20))
for i in range(len(outs)):
ax = fig.add_subplot(2, 2, i + 1)
ax.grid(True, which="both")
outs[i].plot()
plt.show()
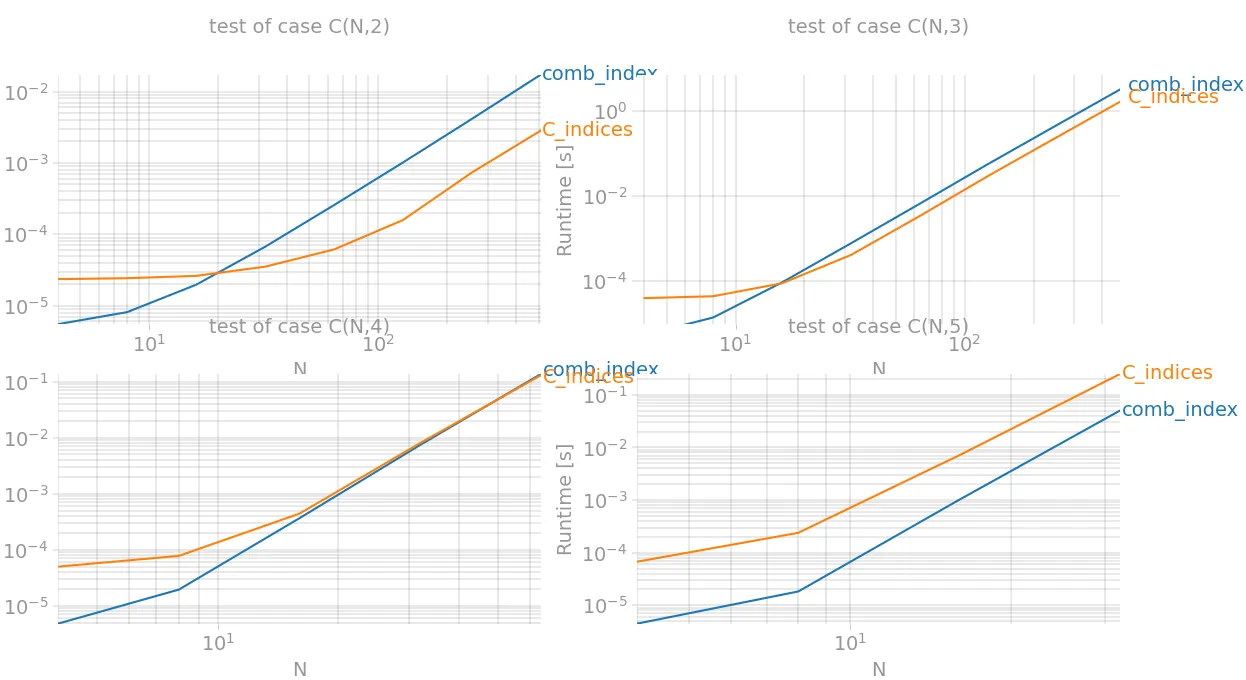
因此,当k=2时(相当于np.triu_indices),性能提升最佳,而当k=3时,速度快了近两倍。
情况k > 3:numpy_combinations(在这里实现)=高达2.5倍的加速
根据此问题(感谢@Divakar),我找到了一种基于前一列和帕斯卡三角形的特定列值计算方式。它还没有被充分优化,但结果非常有前途。我们来看看:
from scipy.linalg import pascal
def stretch(a, k):
l = a.sum()+len(a)*(-k)
out = np.full(l, -1, dtype=int)
out[0] = a[0]-1
idx = (a-k).cumsum()[:-1]
out[idx] = a[1:]-1-k
return out.cumsum()
def numpy_combinations(n, k):
n, k = data
x = np.array([n])
P = pascal(n).astype(int)
C = []
for b in range(k-1,-1,-1):
x = stretch(x, b)
r = P[b][x - b]
C.append(np.repeat(x, r))
return n - 1 - np.array(C).T
以下是基准测试结果:
def build_args(k):
return {'setup': lambda x: (k, x),
'kernels': [comb_index, numpy_combinations],
'n_range': [x for x in range(1, k)],
'xlabel': f'N',
'title': f'test of case C({k}, k)',
'show_progress': True,
'equality_check': False}
outs = [perfplot.bench(**build_args(n)) for n in (12, 15, 17, 23, 25, 28)]
fig = plt.figure(figsize=(20, 20))
for i in range(len(outs)):
ax = fig.add_subplot(2, 3, i + 1)
ax.grid(True, which="both")
outs[i].plot()
plt.show()
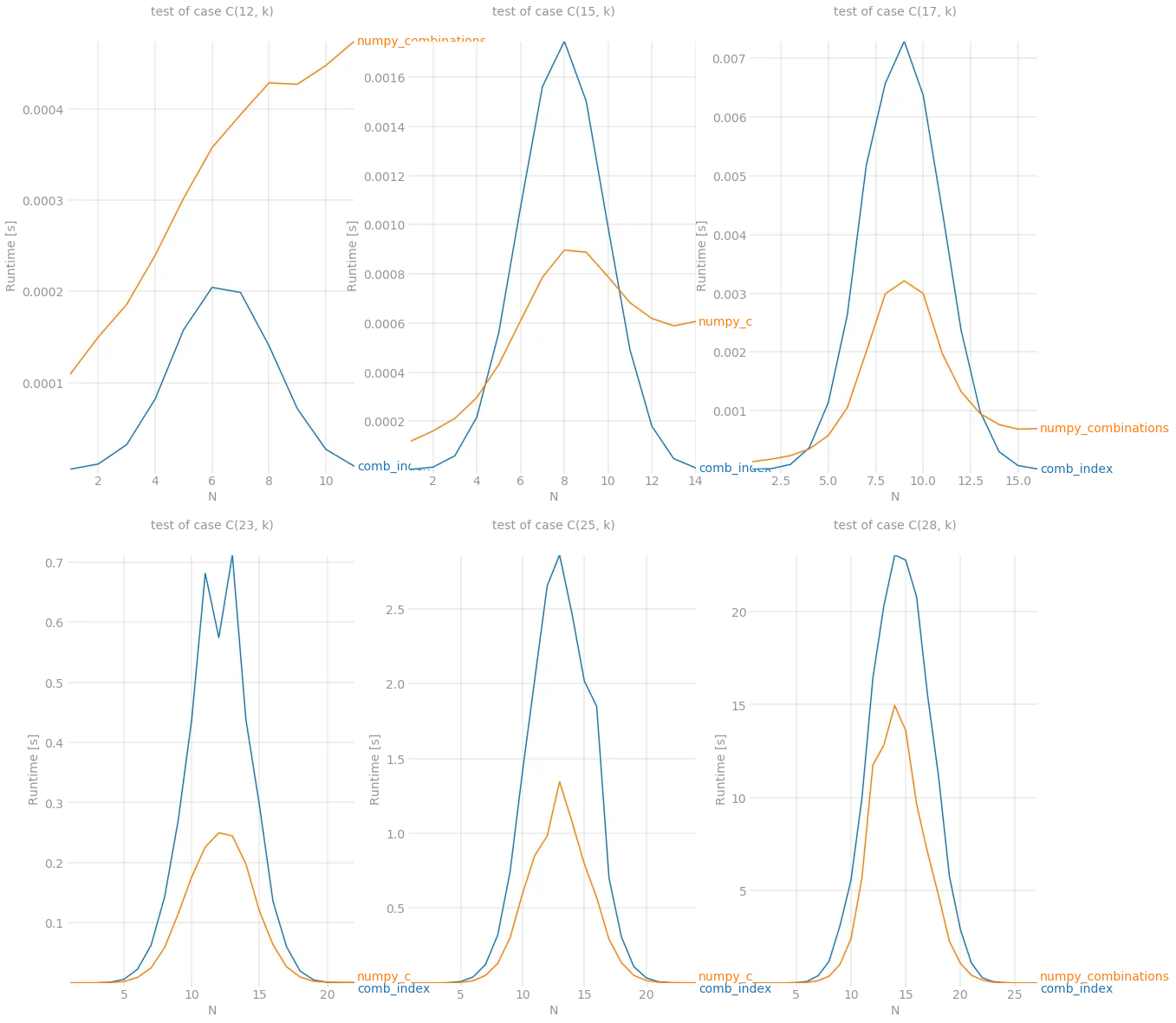
尽管在 n < 15 的情况下仍无法与 itertools.combinations 相抗衡,但在其他情况下,它是新的赢家。最后但并非最不重要的是,当组合数量变得非常大时,numpy 展现了其强大的能力。它能够处理 C(28, 14) 组合,这大约是 14 个元素的 40'000'000 项。



numpy_combinations在获取组合时受限于 n=35,k=3。 - Sengiley