我有一个概率密度函数(PDF)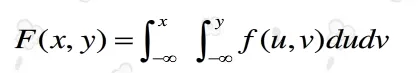 在Scipy中,我可以通过
在Scipy中,我可以通过
现在,我需要整个
主要问题是,对于从
我想知道,由于结果是连续的(例如,您通过
可能有用的链接: 我正在考虑从斐波那契数列借鉴一些东西
f(x,y)。要在点(x,y)处获取其累积分布函数(CDF)F(x,y),您需要对f(x,y)进行积分,如下所示:
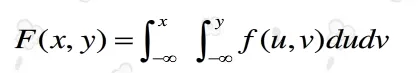 在Scipy中,我可以通过
在Scipy中,我可以通过integrate.nquad来实现:x, y=5, 4
F_at_x_y = integrate.nquad(f, [[-inf, x],[-inf, y]])
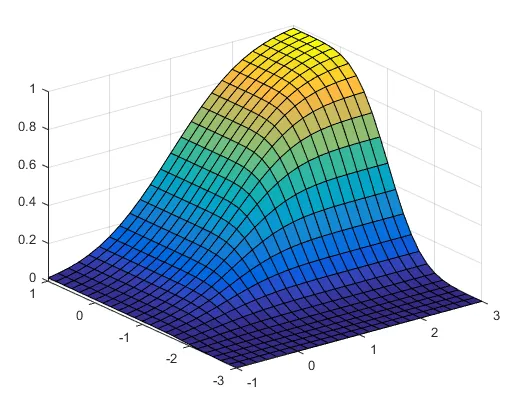
现在,我需要整个
F(x,y) 在 x-y 面板中,类似于这样的东西:
如何做到呢?主要问题是,对于从
(-30,-30) 到 (30,30) 范围内的每一个点,都需要从头开始进行 integrate.nquad 来获取 F(x,y),这太慢了。我想知道,由于结果是连续的(例如,您通过
F(4,4) 的值得到 F(5,6),并积分在这两点之间的区域),是否有可能加速该过程? 因此我们不必在每个点上从头开始运行 integrate ,从而使该过程更快。
可能有用的链接: 我正在考虑从斐波那契数列借鉴一些东西
Ffun=lambda x,y:integrate.nquad(f, [[-inf, x],[-inf, y]]); Fvals=[Ffun(x,y) for x,y in zip(xarr,yarr)],然后按照你所说的,在双重循环中从x[i-1]积分到x[i],并进行比较吗? - Andras Deak -- Слава Україніpdf是作为kdf(http://scikit-learn.org/stable/modules/generated/sklearn.neighbors.KernelDensity.html#sklearn.neighbors.KernelDensity.score_samples) 的瓶颈。multivariate normal的 pdf 比那个快得多。 - ZK Zhao