TL;DR:列表推导式比pdist()快约5倍
from itertools import combinations
from leven import levenshtein
from scipy.spatial.distance import squareform
strings = ["parded", "deputed", "shopbook", "upcheer"]
distances = [levenshtein(i, j) for (i, j) in combinations(strings, 2)]
distance_matrix = squareform(distances)
背景
我对这个问题产生了兴趣,是因为看到一个类似的问题,其中一个答案不能正常工作。
首先,在这个问题中的主要问题是由于pdist()不支持字符串列表,因为它是为数字数据设计的。
Rick's answer很好地解决了这个问题,展示了一种使用Levenshtein包中的距离函数来使用pdist()的方法。然而,正如Tedo Vrbanec在评论中指出的那样,这种方法对于非常大的字符串列表来说速度很慢。应该记住,成对计算的数量按照n(n-1)/2增长,其中n是列表中字符串的数量。
在工作中另一个答案时,我发现可以使用列表推导式和itertools.combinations()来实现相同的结果。我还发现可以通过pool.starmap()使用多处理,而不是列表推导式,希望这样会更快。我进行了以下测试,以找到最快的解决方案。
方法
结果
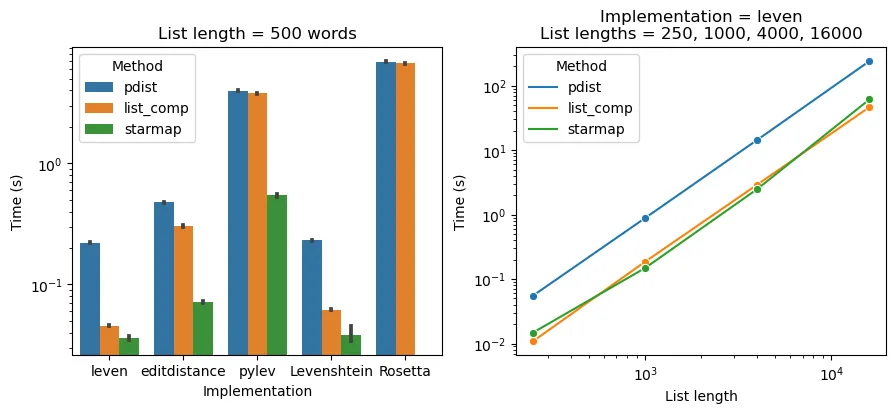
左图显示了计算500个随机抽样单词之间成对距离的平均时间(在五个不同的单词列表上进行平均,误差条为95% CI)。每个条形图都显示了三种方法中的一种(不同颜色),与Levenshtein距离的五种实现之一(x轴)匹配。最右边的绿色条形图缺失,因为Rosetta Code的实现与starmap()不兼容。y轴是对数刻度,以强调最小值之间的差异。
无论使用哪种方法,leven的实现速度最快。虽然starmap()方法通常比列表推导式方法更快,但当两种方法都使用leven的实现时,其优势非常小。我们可能会问这种优势的大小是否取决于单词列表的长度。
在右图中,我将单词列表的长度从250个单词变化到16000个单词,使用leven的实现进行所有测试。对数对数轴上的线性趋势表明,所有三种方法都是字符串对数(n(n-1)/2)的线性。令人惊讶的是,starmap()方法几乎没有比列表推导式方法更大的优势。但是,在所有列表长度上,starmap()和列表推导式方法都比pdist()快约5倍。
结论
计算一组字符串的所有两两Levenshtein距离的最佳方法是在itertools.combinations上使用leven包的距离函数进行列表推导。选择距离函数实现是最具有影响力的因素:请注意这个排名第一的答案推荐了Rosetta Code实现,但它几乎比leven慢100倍。使用starmap()进行进程并行化似乎几乎没有优势,尽管这可能取决于系统。
scikit-learn pairwise_distances()怎么样?
最后,我看到有人建议使用sklearn.metrics.pairwise_distances()或paired_distances(),但我没有运气。据我所知,这些函数需要浮点型数据。尝试将它们用于字符串或字符输入会导致:ValueError: could not convert string to float。
代码
from urllib.request import urlopen
from random import sample
import numpy as np
import pandas as pd
import seaborn as sns
import matplotlib.pyplot as plt
from scipy.spatial.distance import pdist, squareform
from time import time
from multiprocessing import Pool, cpu_count
from itertools import combinations
url = "https://raw.githubusercontent.com/dwyl/english-words/master/words_alpha.txt"
all_words = urlopen(url).read().splitlines()
import leven
import editdistance
import pylev
import Levenshtein
def levenshteinDistance(str1, str2):
m = len(str1)
n = len(str2)
d = [[i] for i in range(1, m + 1)]
d.insert(0, list(range(0, n + 1)))
for j in range(1, n + 1):
for i in range(1, m + 1):
if str1[i - 1] == str2[j - 1]:
substitutionCost = 0
else:
substitutionCost = 1
d[i].insert(
j,
min(
d[i - 1][j] + 1, d[i][j - 1] + 1, d[i - 1][j - 1] + substitutionCost
),
)
return d[-1][-1]
lev_implementations = [
leven.levenshtein,
editdistance.eval,
pylev.wfi_levenshtein,
Levenshtein.distance,
levenshteinDistance,
]
lev_impl_names = {
"levenshtein": "leven",
"eval": "editdistance",
"wfi_levenshtein": "pylev",
"distance": "Levenshtein",
"levenshteinDistance": "Rosetta",
}
def pdist_(strings, levenshtein):
transformed_strings = np.array(strings).reshape(-1, 1)
return pdist(transformed_strings, lambda x, y: levenshtein(x[0], y[0]))
def list_comp(strings, levenshtein):
return [levenshtein(i, j) for (i, j) in combinations(strings, 2)]
def starmap(strings, levenshtein):
return pool.starmap(levenshtein, combinations(strings, 2))
methods = [pdist_,list_comp,starmap]
pool = Pool(processes=cpu_count())
N_sims = 5
N_words = 500
times = []
impls = []
meths = []
for simulations in range(N_sims):
strings = [x.decode() for x in sample(all_words, N_words)]
for method in methods:
for levenshtein in lev_implementations:
if (method == starmap) & (levenshtein == levenshteinDistance):
continue
t0 = time()
distance_matrix = method(strings, levenshtein)
t1 = time()
times.append(t1 - t0)
meths.append(method.__name__.rstrip("_"))
impls.append(lev_impl_names[levenshtein.__name__])
df = pd.DataFrame({"Time (s)": times, "Implementation": impls, "Method": meths})
word_counts = [250, 1000, 4000, 16000]
pool = Pool(processes=cpu_count())
N_sims = 1
times = []
meths = []
comps = []
ll = []
for simulations in range(N_sims):
strings_multi = {}
for N in word_counts:
strings = [x.decode() for x in sample(all_words, N)]
for method in methods:
t0 = time()
distance_matrix = method(strings, leven.levenshtein)
t1 = time()
times.append(t1 - t0)
meths.append(method.__name__.rstrip("_"))
comps.append(sum([1 for _ in combinations(strings, 2)]))
ll.append(N)
df2 = pd.DataFrame({"Time (s)": times, "Method": meths, "Number of string pairs": comps, "List length": ll})
fig, axes = plt.subplots(1, 2, figsize=(10.5,4))
sns.barplot(x="Implementation", y="Time (s)", hue="Method", data=df, ax=axes[0])
axes[0].set_yscale('log')
axes[0].set_title('List length = %i words' % (N_words,))
sns.lineplot(x="List length", y="Time (s)", hue="Method", data=df2, marker='o', ax=axes[1])
axes[1].set_yscale('log')
axes[1].set_xscale('log')
axes[1].set_title('Implementation = leven\nList lengths = 250, 1000, 4000, 16000')
