我正在尝试使用{{link1:
我很难将二维数据集放入
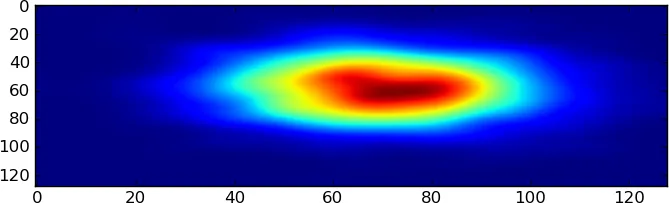
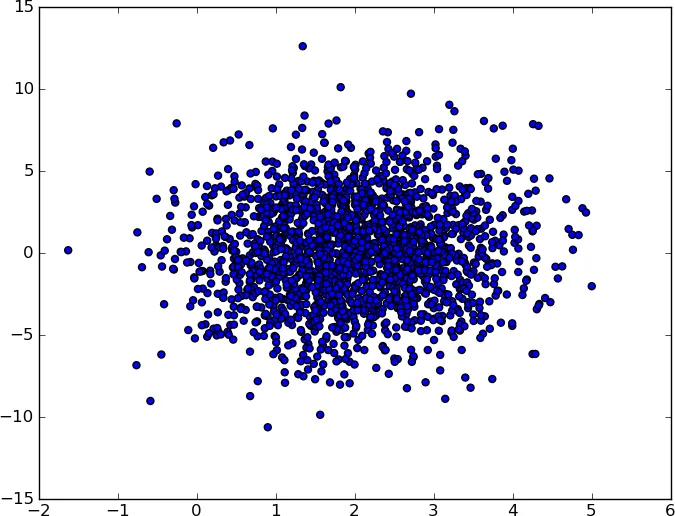
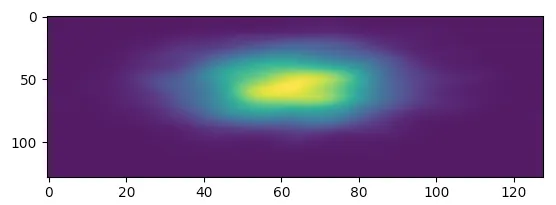
这段话的意思是:我有三个点,坐标分别为[1.1, 1.1]、[1.2, 1.2]和[1.3, 1.3]。我想使用x轴和y轴宽度为1来进行核密度估计,从1到3进行估计。
创建高斯核密度时,一直出现错误。
scipy.stats.gaussian_kde类}}来平滑一些带有纬度和经度信息的离散数据,以便最终显示为类似于等高线地图的形式,其中高密度是峰值,低密度是山谷。我很难将二维数据集放入
gaussian_kde类中。我已经尝试了一维数据的操作,所以我认为二维数据应该是这样的:from scipy import stats
from numpy import array
data = array([[1.1, 1.1],
[1.2, 1.2],
[1.3, 1.3]])
kde = stats.gaussian_kde(data)
kde.evaluate([1,2,3],[1,2,3])
这段话的意思是:我有三个点,坐标分别为[1.1, 1.1]、[1.2, 1.2]和[1.3, 1.3]。我想使用x轴和y轴宽度为1来进行核密度估计,从1到3进行估计。
创建高斯核密度时,一直出现错误。
raise LinAlgError("singular matrix")
numpy.linalg.linalg.LinAlgError: singular matrix
查看gaussian_kde的源代码后,我意识到我的数据集解释与计算维数方式完全不同,但我找不到任何示例代码展示该模块如何处理多维数据。有人能提供一些使用gaussian_kde处理多维数据的示例方法吗?