有人知道在Python中计算经验/样本协方差图的好方法吗?
这是一个书籍的截图,其中包含了协方差图的良好定义:

首先,我是否理解正确?如果是这样,假设在二维空间中,有什么好的方法来计算这个问题呢?我试图用Python编写代码(使用numpy和pandas),但需要几秒钟时间,而且我甚至不确定它是否正确,这就是为什么我会避免在此处发布代码的原因。以下是另一种非常天真的实现尝试:
from scipy.spatial.distance import pdist, squareform
distances = squareform(pdist(np.array(coordinates))) # coordinates is a nx2 array
z = np.array(z) # z are the values
cutoff = np.max(distances)/3.0 # somewhat arbitrary cutoff
width = cutoff/15.0
widths = np.arange(0, cutoff + width, width)
Z = []
Cov = []
for w in np.arange(len(widths)-1): # for each width
# for each pairwise distance
for i in np.arange(distances.shape[0]):
for j in np.arange(distances.shape[1]):
if distances[i, j] <= widths[w+1] and distances[i, j] > widths[w]:
m1 = []
m2 = []
# when a distance is within a given width, calculate the means of
# the points involved
for x in np.arange(distances.shape[1]):
if distances[i,x] <= widths[w+1] and distances[i, x] > widths[w]:
m1.append(z[x])
for y in np.arange(distances.shape[1]):
if distances[j,y] <= widths[w+1] and distances[j, y] > widths[w]:
m2.append(z[y])
mean_m1 = np.array(m1).mean()
mean_m2 = np.array(m2).mean()
Z.append(z[i]*z[j] - mean_m1*mean_m2)
Z_mean = np.array(Z).mean() # calculate covariogram for width w
Cov.append(Z_mean) # collect covariances for all widths
然而,现在我已经确认我的代码存在错误。我知道这是因为我使用变程图来计算协方差函数(协方差函数(h) = 协方差函数(0) - 变程图(h)),并且得到了不同的图形:
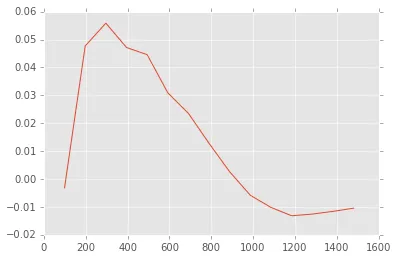
并且它应该看起来像这样:
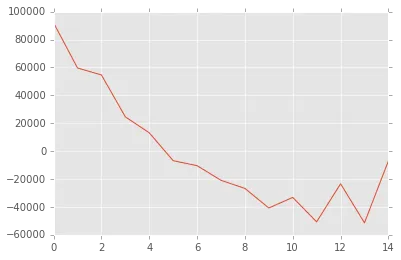
最后,如果您知道一个Python/R/MATLAB库来计算经验协变函数,请告诉我。至少这样我可以验证我所做的。
m方程没有意义。如果你对i求和,那么在求和之外用i作为索引就没有任何意义(例如,在m(x_i)中);也就是说,右侧没有i。 - tom10