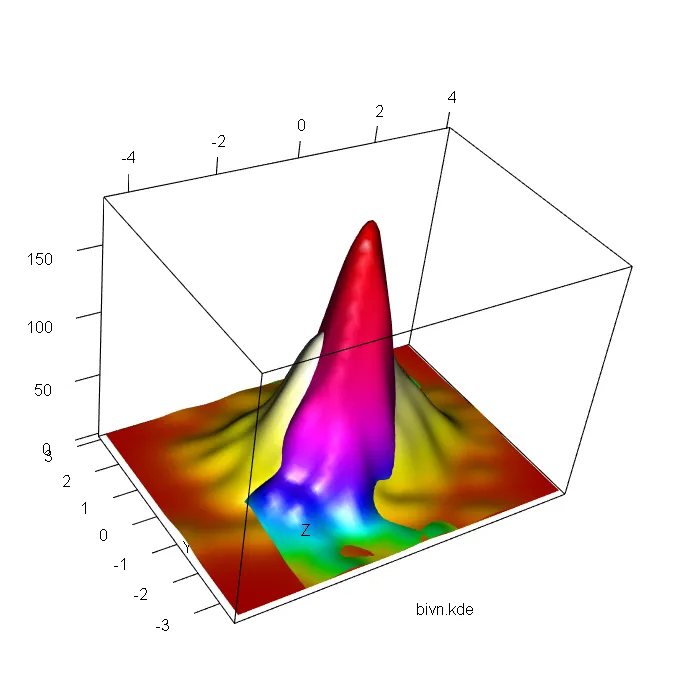
我一直在使用 MASS 软件包进行操作,例如可以使用 image 和 par(new=TRUE) 绘制两个双变量正态分布图:
# lets first simulate a bivariate normal sample
library(MASS)
bivn <- mvrnorm(1000, mu = c(0, 0), Sigma = matrix(c(1, .5, .5, 1), 2))
bivn2 <- mvrnorm(1000, mu = c(0, 0), Sigma = matrix(c(1.5, 1.5, 1.5, 1.5), 2))
# now we do a kernel density estimate
bivn.kde <- kde2d(bivn[,1], bivn[,2], n = 50)
bivn.kde2 <- kde2d(bivn2[,1], bivn[,2], n = 50)
# fancy perspective
persp(bivn.kde, phi = 45, theta = 30, shade = .1, border = NA)
par(new=TRUE)
persp(bivn.kde2, phi = 45, theta = 30, shade = .1, border = NA)
这样看起来并不太好,我想我只能调整坐标轴和其他内容。但是,如果我尝试使用相似的方法来处理等高线图,它们并不会重叠。它们只是被替换了:
# fancy contour with image
image(bivn.kde); contour(bivn.kde, add = T)
par(new=TRUE)
image(bivn.kde2); contour(bivn.kde, add = T)
这是我想要的最佳方法吗?还是我只是自己把事情搞得很难? 欢迎任何建议。谢谢!